ARTÍCULO |
Estudio de asociación epigenómica en la pérdida de peso tras una intervención nutricional: Estudio RESMENA
María Luisa Mansego, Marian Zulet, Javier Campión J, Fermín Milagro F, José Alfredo Martínez*
CIBER CB06/03 Fisiopatología de la Obesidad y la Nutrición, (CIBEROBN), Instituto de salud Carlos III, Madrid, EspaĖa y Centro de Investigación en Nutrición y Departamento de Ciencias de la Alimentación y Fisiología, Universidad de Navarra. C/ Irunlarrea 1, Pamplona, Navarra 31008, EspaĖa
*e-mail: jalfmtz@unav.es
Recibido el 8 de abril de 2014 An. Real Acad. Farm. Vol. 80, Nľ 3 (2014), pag.614-623
resumen
Mediante una aproximación epigenómica, se analizaron las posibles asociaciones entre los niveles basales en la metilación del ADN y una mejor respuesta a la pérdida de peso después de un programa de intervención nutricional en la población obesa del estudio RESMENA. Esta investigación ha identificado 3 regiones de ADN (genes RGS6, A2BP1 y RASGRF1) que se encuentran diferencialmente metiladas entre sujetos con alta y baja respuesta a la pérdida de peso. Además, estos genes están implicados en la misma ruta metabólica y habían sido previamente significativamente asociados con la obesidad. |
Palabras clave: Metilación del ADN; intervención nutricional; pérdida de peso.
abstract
Epigenomic association study in weight loss after a nutritional intervention: RESMENA Study
Through anepigenomics approach, the possible association between baseline levels in DNA methylation and a better weight loss response after a multidisciplinary intervention program were analyzedin obese population from RESMENA-S study. Three DNA regions that are differentially methylated (RGS6, A2BP1 and RASGRF1 genes) showed differential methylation levels at baseline between high and low responders to the multidisciplinary weight loss intervention. Moreover, these genes were implicated in the same metabolic pathway and have been previously significantly associated with obesity. |
Keywords: DNA methylation; nutritional intervention; weight loss.
1. INTRODUCcióN
La obesidad está considerada como uno de los principales problemas de salud en la mayoría de los países por su creciente incidencia en la población y por los trastornos fisiopatológicos asociados (7). La obesidad común es el resultado de la interacción entre factores ambientales y de comportamiento, como la dieta y el ejercicio, con componentes genéticos específicos de cada individuo (17). Así, la predisposición genética es un elemento importante en el desarrollo de obesidad, por lo que las variaciones genotípicas en determinados nucleótidos (SNPs) relacionados con la obesidad son, sin duda, de gran relevancia en dicha susceptibilidad (5). Por otra parte, los procesos epigenéticos tienen un papel clave en la regulación de la expresión génica, modulando así la predisposición génica dependiente de la secuencia de nucleótidos. En este sentido, diversas investigaciones han revelado que diversos mecanismos epigenéticos durante las etapas perinatales (intrauterina, lactancia,…), como el estrés o la dieta materna, están implicados en el desarrollo posterior de obesidad y que, asimismo, la dieta y otros factores ambientales que tienen lugar durante la etapa adulta también pueden influir en la acumulación de grasa a través de mecanismos epigenéticos (3). Incluso, algunas de dichas modificaciones epigenéticas parecen ser transmitidas a la siguiente generación a través de los gametos, por lo que son un serio candidato a explicar una parte del imparable incremento de la prevalencia de obesidad entre niĖos y adolescentes (21). De hecho, diversos estudios apuntan hacia la importancia de las diferencias epigenéticas entre los diferentes individuos con mayor o menor susceptibilidad al desarrollo de obesidad y otras comorbilidades asociadas o a la facilidad para la pérdida de peso o su mantenimiento posterior (2, 18).
Los principales procesos epigenéticos que podrían influir en el desarrollo de obesidad, debido a su papel en la regulación de la expresión de genes relacionados con ella, son la metilación del ADN en las islas CpG, las modificaciones covalentes en las colas de las histonas por metilación, acetilación o ubiquitinación, y las proteínas reguladoras que mantienen la cromatina en un estado activo (trithorax) o silencioso (polycombs) (6). Algunos autores incluyen también los microRNAs (miRNA) como elementos de regulación epigenética, bien por controlar la expresión de importantes genes reguladores de la epigenética como las acetilasas de histonas o las DNA metiltransferasas, como por el control epigenético de la expresión de ciertos miRNAs(23).
Algunas alteraciones metabólicas asociadas con la obesidad, como la hiperglucemia y la resistencia a la insulina, así como la presencia de factores prometilantes en la dieta, como el ácido fólico o la metionina, parecen influir de manera decisiva sobre los procesos epigenéticos que afectan a genes relacionados con la obesidad (4). Además, estos mecanismos epigenéticos están influenciados por otros factores habitualmente asociados a la obesidad, incluyendo la inflamación, a través de la acción de citoquinas proinflamatorias como TNFα e IL-6 (27), el estrés oxidativo (9) y las condiciones de hipoxia que se desarrollan en el tejido adiposo obeso (26). Todas estas modificaciones pueden ser afectadas por diversos fenómenos ambientales, incluyendo numerosos factores dietéticos, y se ha sugerido que se podrían transmitir por un tipo de herencia epigenética transgeneracional (20). En este sentido, un estudio preliminar (19) ha vinculado la obesidad en ratas alimentadas con una dieta rica en grasa durante la edad adulta con una mayor metilación del promotor de la leptina. Asimismo, se ha comprobado que dicha dieta es capaz de alterar la metilación de genes tan importantes como la ácido graso sintasa (FAS) o el gen mitocondrial NDUFB6(15).
Por lo tanto, nuestro objetivo fue investigar los posibles cambios en los patrones de metilación de ADN entre los individuos que mostraron una alta o baja respuesta a un programa integral de pérdida de peso en obesos.
2. material y métodos
Población de estudio y diseĖo experimental
El estudio de la reducción del síndrome metabólico en Navarra-EspaĖa (RESMENA-S) es una estrategia multidisciplinar basada en la crononutrición y la educación nutricional, junto con control dietético y psicológico, que consistió en un ensayo aleatorizado de diseĖo paralelo y prospectivo en el que participaron un total de 100 individuos. (28). El grupo RESMENA-S (n = 50) siguió una dieta personalizada de pérdida de peso (restricción energética 30%), con una distribución en macronutrientes (hidratos de carbono/grasas/ proteínas) de 40/30/30, elevada frecuencia de ingestas (7/día), bajo índice/ carga glucémica y elevada capacidad antioxidante y adherencia a la dieta Mediterránea (28). El grupo control (n = 50) siguió una dieta con la misma restricción energética y basada en la Asociación Americana del Corazón (AHA). El estudio tuvo una duración de 8 semanas bajo control dietético y psicológico en ambos grupos. Durante un periodo adicional de 16 semanas de auto-control, los voluntarios siguieron el mismo patrón dietético pero sin ningún asesoramiento específico.
El estudio de los niveles de metilación se llevó a cabo en una submuestra de 46 sujetos obesos (50% varones). Estos participantes fueron seleccionados de la muestra de población general y eran los que tuvieron una mejor o peor respuesta a la intervención de pérdida de peso. Se les consideró como "alta respuesta" (perder más de un 5% de su peso inicial al cabo de 24 semanas de intervención; n=26) y "baja respuesta" (aquellos que no lograron una pérdida de su peso del 5%; n=17), respectivamente.
Todos los sujetos dieron su consentimiento informado por escrito (http://www.clinicaltrials.gov; NCT01087086), el estudio fue aprobado por el Comité de Ética de la Universidad de Navarra (065/2009) y estuvo de acuerdo con la Declaración de Helsinki (revisada en Corea del Sur en 2008).
Evaluaciones antropométricas y bioquímicas
Las medidas antropométricas (peso, talla, circunferencia de cintura y cadera) se llevaron a cabo con los sujetos en ropa interior. El peso corporal se midió con una precisión de 0,1 kg con una Tanita SC-330, (Tanita Corp, Japón). La talla se estimó con un tallímetro (Seca 713 modelo, Postfach, Alemania) con una precisión de 1 mm. El índice de masa corporal (IMC) se calculó como el peso corporal dividido por la altura al cuadrado (kg/m2). Las circunferencias de la cintura y la cadera se midieron siguiendo protocolos validados (28). La grasa corporal total se midió por impedancia bioeléctrica Tanita SC-330 (TanitaCorp, Japón) y por absorciometría con rayos X de doble energía (DXA) (Prodigy, versión de software 6,0, Madison, WI) como se describió previamente(28).
Las concentraciones séricas de glucosa, colesterol total, lipoproteínas de alta densidad-colesterol (HDL-c) y triglicéridos se midieron en un autoanalizador Pentra C-200 (HORIBA ABX, Madrid, EspaĖa) con los kits específicos. Las concentraciones de insulina se determinaron mediante un kit de ensayo inmunoenzimático (Mercodia, Uppsala, Suecia) en un autoanalizador Triturus (Grifols SA, Barcelona, EspaĖa).
Extracción de ADN
Muestras de sangre venosa se recogieron al inicio del estudio para la obtención de ADN. El ADN genómico fue extraído utilizando el kit de purificación de ADN Master Purefor Blood Version II (Biotecnologías Epicenter, Madison, WI, EE.UU.) y se almacenó a -80°C hasta su procesamiento.
Determinación de patrones de metilación de ADN
El perfil epigenético del ADN genómico fue analizado en 48 individuos seleccionados del estudio RESMENA-S utilizando el ensayo de metilación Human Methylation 450 BeadChip (Illumina, San Diego, CA) conteniendo más de 485.000 sitios CpGs individuales. Brevemente, 500 ng de ADN genómico tratado con bisulfito sódico (EZ DNA Methylation™ Kit; Epitect, Qiagen) fue amplificado, marcado, hibridado con sondas metiladas y no metiladas y escaneado de los microarrays en la plataforma HiScanSQ de Illumina, Inc. El análisis de imagen y determinación de la seĖal de las sondas se realizó usando el software Genome Studio, Módulo de metilación (Illumina, Inc).
Los datos de metilación en un sitio CpG concreto se obtuvieron a partir de las intensidades de seĖal para la sonda metilada (M) y la no metilada (U) para ADN genómico en esa posición. Así, los nivel de metilación (β) fueron calculados como la proporción de metilado respecto al total de la seĖal [i.e., β = M/(M + U)], donde el rango β oscila desde 0 (no metilado) a 1 (metilado).
El análisis de las rutas metabólicas se realizó con el uso de Ingenuity Pathway Analysis (IPA; Ingenuity Systems, Inc., Redwood City, CA, EE.UU.; http://www.ingenuity.com), que incluye datos de forma manual precisos y trazables totalmente derivados de fuentes bibliográficas.
Análisis estadísticos
Los análisis estadísticos se realizaron utilizando el paquete estadístico SPSS 15.0 (SPSS, Chicago, IL, EE.UU.). La prueba de Kolmogorov-Smirnov y Shapiro-Wilks se utilizaron para determinar la normalidad en la distribución variable.
Las diferencias en los parámetros clínicos y antropométricos tras la intervención entre respondedores altos y bajos a la dieta fueron evaluadas con la prueba t-Student o la U de Mann Whitney. El gráfico de volcán y la diferencia entre los distintos sitios CpGs fueron evaluados por ANOVA de una vía (respuesta al efecto de la dieta ajustado por edad y efecto del uso de distintos microarrays) gracias al programa Array Studio 5.0 (Omicsoft, Cary, NC, USA). Se aplicó la tasa de falso descubrimiento (False DiscoverRate; FDR) en los ajustes por comparaciones múltiples.
3. resultados y discusión
El presente proyecto se ha centrado en la selección de los individuos que participaron en el estudio RESMENA-S hasta el final del mismo. Las características generales de los participantes agrupados en función de su respuesta a la intervención dietética, no difirieron significativamente entre los sujetos del grupo control y RESMENA-S en variables antropométricas, presión arterial y perfil lipídico (Tabla 1). Sin embargo, los individuos con una alta respuesta a la dieta mostraron una reducción significativa en la mayoría de los parámetros antropométricos con respecto a sus valores iniciales de los mismos.
Con respecto a los resultados obtenidos en el microarray Human Methylation450 BeadChip, tras analizar los controles de calidad de dicho array se redujo la muestra a 46 sujetos analizados. El gráfico de volcán mostró la distribución de los 485512 sitios CpG analizados con respecto a la respuesta frente a la pérdida de peso debida a la dieta (Figura 1). El estudio de microarray mostró 90 sitios CpG diferencialmente metilados (P-valor <0,01) entre sujetos con alta respuesta a la dieta frente a los que presentaron una baja respuesta a la intervención nutricional. Ninguno de estos CpG fue estadísticamente significativo tras los ajustes por comparaciones múltiples aplicando FDR. Cuarenta de estos sitios se encontraron hipometilatos y cincuenta hipermetilados en el grupo de los sujetos con baja respuesta comparado con los que mostraron alta respuesta a la perdida de peso. Las características de los 10 principales genes asociados independientemente con la respuesta a la dieta (Tabla 2).
Tabla 1.- Características clínicas de 46 obesos en situación basal y tras 24 semanas de intervención dietética de la población RESMENA-S.
Variables |
Alta respuesta (N=28) |
Baja respuesta (N=20) |
||
Basal |
Después de 24 semanas |
Basal |
Después de 24 semanas |
|
Sexo (N mujeres, %) |
14 (48,9) |
9 (47,9) |
||
Edad (aĖos) |
49,9 (10,6) |
45,6 (8,2) |
||
Tipo de dieta (N control(%)) |
11 (41,4) |
9 (47,9) |
||
Peso (kg) |
100,9 (17,6) |
90,0 (18,6) * |
105,2 (18,2) |
108,0 (17,5) |
IMC (kg/m2) |
35,9 (4,8) |
31,9 (4,9) ** |
36,9 (1,9) |
35,9 (2,0) |
Cintura (cm) |
110,9 (13,3) |
101,3 (14,9) * |
114,8 (11,9) |
114,9 (9,2) |
Cadera (cm) |
116,2 (9,3) |
109,1 (9,4) ** |
118,9 (7,1) |
116,7 (7,9) |
Índice CC |
1,0 (0,1) |
0,9 (0,1) |
1,0 (0,1) |
1,0 (0,1) |
PAS (mmHg) |
151,3 (18,1) |
131,2 (13,6)** |
145,9 (16,2) |
140,3 (14,3) |
PAD (mmHg) |
85,3 (9,8) |
75,8 (10,0) ** |
85,6 (6,4) |
81,9 (11,4) |
Masa grasa (%) |
38,1 (8,1) |
33,2 (8,0) * |
41,2 (5,5) |
37,1 (6,2) |
Glucosa (mg/dl) |
117,2 (28,1) |
104,0 (16,2) * |
133,4 (43,7) |
128,3 (34,0) |
CT (mg/dl) |
221,2 (51,1) |
213,1 (41,1) |
216,3 (50,3) |
215,2 (52,7) |
HDLc (mg/dl) |
44,2 (10,1) |
47,6 (11,2) |
41,2 (11,5) |
47,3 (15,5) |
TG (mg/dl) |
178,5 (86,5) |
123,4 (41,9)* |
220,6 (142,3) |
229,3 (163,3) |
Insulina (mUl) |
13,1 (7,5) |
6,3 (3,8) ** |
18,9 (11,3) |
16,0 (10,6) |
Media (Desviación estándar). IMC: Índice de Masa Corporal; Índice CC: Índice cintura/cadera; PAS: Presión arterial sistólica; PAD: Presión arterial diastólica; CT: Colesterol total; HDLc: lipoproteínas de alta densidad-colesterol; TG: Triglíceridos. *P-valor <0.05, **P-valor <0,001 frente a los valores basales.
Con respecto a los resultados de IPA sobre rutas metabólicas implicadas para estos 90 sitios CpGs diferencialmente metilados entre los sujetos con alta y baja respuesta tras la intervención, se identificaron redes relacionadas con el cáncer, las enfermedades del sistema reproductivo y la respuesta inflamatoria, ciclo celular, lesiones organismo, y las anomalías y cáncer, y el movimiento celular, el tráfico de célula inmune, desarrollo y función del sistema hematológico que participan en la respuesta de la pérdida de peso (Figura 2).
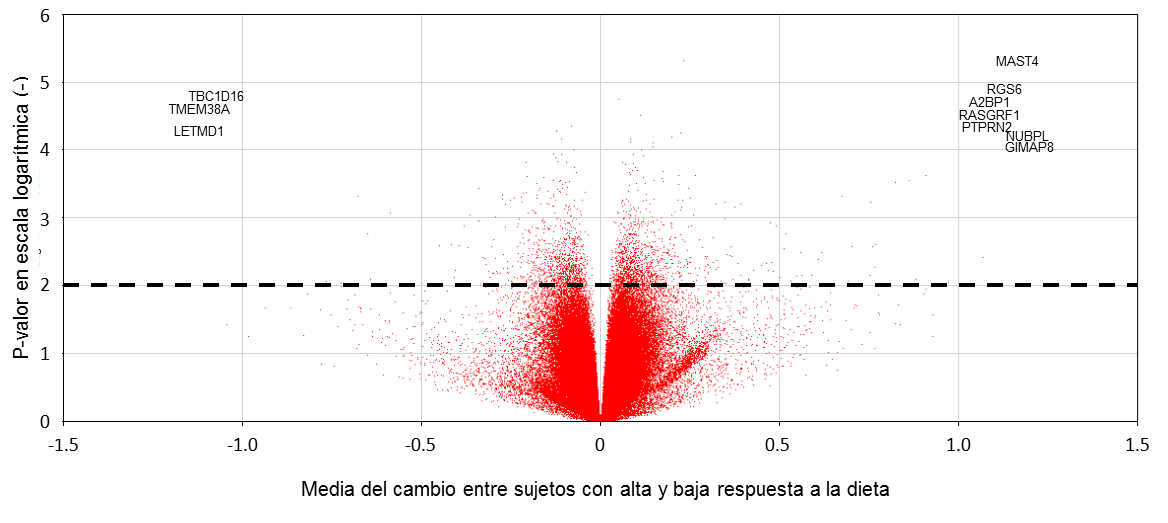
Figura 1.- Gráfico tipo volcán de la asociación de metilación ajustado por edad y sexo (N=43) La línea discontinua muestra los genes con niveles de metilación significativamente diferenciados entre sujetos con alta y baja respuesta a la dieta (p-valor < 0.01).
Tabla 2.-Características genéticas de los 10 CpGs con mayor asociación a la respuesta dietética ajustados por edad y sexo en 43 individuos.
Gen |
ID Illumina |
Cr |
Posición CpG |
Media de cambio |
P-valor |
MAST4 |
cg01933079 |
5 |
65929713 |
1.174 |
5.02E-06 |
RGS6 |
cg10741333 |
14 |
72400168 |
1.0797 |
3.07E-05 |
TBC1D16 |
cg16775460 |
17 |
77984481 |
-1.0581 |
4.41E-05 |
TMEM38A |
cg04172000 |
19 |
16771088 |
-1.0879 |
5.34E-05 |
A2BP1 |
cg01050225 |
16 |
7703947 |
1.1481 |
6.34E-05 |
RASGRF1 |
cg15156078 |
15 |
79383413 |
1.0601 |
6.86E-05 |
LETMD1 |
cg01016169 |
12 |
51441443 |
-1.0777 |
6.93E-05 |
PTPRN2 |
cg25029446 |
7 |
157495282 |
1.0594 |
7.70E-05 |
NUBPL |
cg22905571 |
14 |
32269554 |
1.1076 |
7.90E-05 |
GIMAP8 |
cg01364581 |
7 |
150175800 |
1.108 |
8.80E-05 |
ID:Identificador, Cr: cromosoma. Version 37.
El reciente concepto "nutrición personalizada" asociado a los campos de la genómica, la proteómica y la metabolómica está surgiendo con fuerza en los últimos tiempos (16). En ese sentido, los estudios genómicos se están dirigiendo hacia la prevención de la obesidad basándose en dietas personalizadas (22). En consecuencia, los marcadores genéticos y epigenéticos han sido considerados como herramientas de predicción para evaluar las diferencias individuales en la capacidad de respuesta después de una intervención nutricional (12). Nuestro análisis reveló que el patrón de la metilación epigenética diferencial entre sujetos con alta y baja respuesta a un programa de pérdida de peso multidisciplinario podría ayudar a predecir la disminución de peso en obesos adultos.
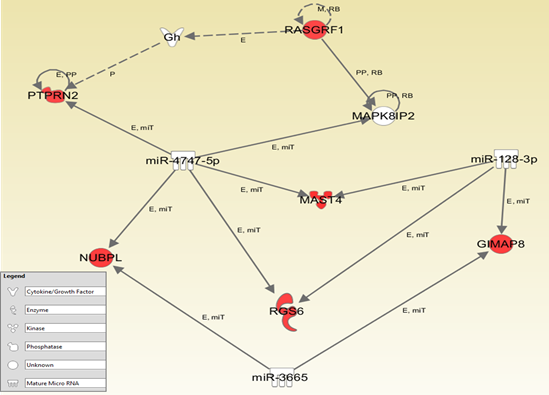
Figura 2.- Ruta metabólica cuyas funciones principales son la seĖalización y morfología celular y compuesta por 6 de los genes con mayor asociación a la respuesta de la dieta (RGS6, PTPRN2, MAST4, RASGRF1, GIMAP8, NUBPL).
Adicionalmente, se han identificado algunos genes con una clara influencia sobre la obesidad como es el caso del gen A2BP1 (cg01050225), que codifica para la proteína 1 de unión a ataxina-2 (también conocido como Fox-1) y puede estar implicado en transcripción alternativa de tejidos específicos (11). Además, este gen contiene motivos de unión a ARN, A2BP1 también interactúa con la ataxina-2 (ATXN2) (24), una proteína que parece ser que participan en el metabolismo de ARN (14). ATXN2 ha sido relacionada en la enfermedad neurodegenerativa, ataxia espinocerebelosa tipo 2 (SCA2) (14), y la hiperfagia y la obesidad son dos características clínicas principales en SCA2 (1). Consistente con esta observación, se ha descrito como ratones knockout para ATXN2 (Sca-/ -) presentan mayores valores de IMC que ratón tipo salvaje para este gen cuando ambos son alimentados con una dieta alta en grasas (10, 13).
Otro de los genes asociados a la respuesta dietética es el RGS6 que a su vez se ha descrito como gen candidato para el estudio de la adiposidad, ya que pueden estar asociados con una tendencia conductual hacia el consumo de alimentos cargado de grasa (25).
Por último, una asociación entre la metilación génica del gen RASGRF1 al inicio del estudio y los cambios antropométricos después de la intervención. Los sujetos que respondieron eficazmente al tratamiento mostraron porcentajes de metilación significativamente más altos después del tratamiento.RASGRF1 está implicado en el mantenimiento de la homeostasis de la glucosa mediante la regulación de las células beta pancreáticas (8). Ratones knock-out para el gen RASGRF1 muestran un fenotipo similar a las manifestaciones de tipo 2 de diabetes preclínica (8).
En resumen, este trabajo describe un patrón epigenético diferencial en sujetos de la población RESMENAS que podría ser aplicado para predecir cambios de peso corporal. También se ha identificado 90 regiones de ADN diferencialmente metiladas, dependiendo de la respuesta de la pérdida de peso. Estos cambios epigenéticos pueden ayudar a entender y predecir mejor la respuesta para perder peso.
4. agradecimientos
Este trabajo fue apoyado por Línea Especial sobre Nutrición, Obesidad y Salud (Universidad de Navarra LE/97), la fundación Mapfre y CIBER CB06/03 Fisiopatología de la Obesidad y la Nutrición, (CIBEROBN), Instituto de salud Carlos III. A M.L.M. se le fue concedida una beca Juan de la Cierva del Ministerio de Economía y Competitividad del gobierno de EspaĖa.
5. referencias
1. Abdel-Aleem, A; Zaki, MS. Spinocerebellar ataxia type 2 (SCA2) in an Egyptian family presenting with polyphagia and marked CAG expansion in infancy. J Neurol 255, 413-419(2008).
11. Kuroyanagi, H. Fox-1 family of RNA-binding proteins. Cell Mol Life Sci 66, 3895-3907(2009).
14. Lastres-Becker, I; Rub, U; Auburger, G. Spinocerebellar ataxia 2 (SCA2). Cerebellum 7, 115-124 (2008).
15. Lomba, A; Martinez, JA; Garcia-Diaz, DF; Paternain, L; Marti, A; Campion, J; Milagro, FI. Weight gain induced by an isocaloric pair-fed high fat diet: a nutriepigenetic study on FASN and NDUFB6 gene promoters. Mol Genet Metab 101, 273-278(2010).
16. Marti, A; Goyenechea, E; Martinez, JA. Nutrigenetics: a tool to provide personalized nutritional therapy to the obese. J Nutrigenet Nutrigenomics 3, 157-169 (2010).
17. Martinez, JA; Parra, MD; Santos, JL; Moreno-Aliaga, MJ; Marti, A; Martinez-Gonzalez, MA. Genotype-dependent response to energy-restricted diets in obese subjects: towards personalized nutrition. Asia Pac J Clin Nutr 17 Suppl 1, 119-122(2008).
18. Milagro, FI; Campion, J; Cordero, P; Goyenechea, E; Gomez-Uriz, AM; Abete, I; Zulet, MA; Martinez, JA. A dual epigenomic approach for the search of obesity biomarkers: DNA methylation in relation to diet-induced weight loss. FASEB J 25, 1378-1389 (2011).
19. Milagro, FI; Campion, J; Garcia-Diaz, DF; Goyenechea, E; Paternain, L; Martinez, JA. High fat diet-induced obesity modifies the methylation pattern of leptin promoter in rats. J Physiol Biochem 65, 1-9(2009).
20. Ng, SF; Lin, RC; Laybutt, DR; Barres, R; Owens, JA; Morris, MJ. Chronic high-fat diet in fathers programs beta-cell dysfunction in female rat offspring. Nature 467, 963-966 (2010).
22. Razquin, C; Marti, A; Martinez, JA. Evidences on three relevant obesogenes: MC4R, FTO and PPARgamma. Approaches for personalized nutrition. Mol Nutr Food Res 55, 136-149(2011).
28. Zulet, MA; Bondia-Pons, I; Abete, I; de la Iglesia, R; Lopez-Legarrea; P, Forga, L; Navas-Carretero, S; Martinez, JA. The reduction of the metabolyc syndrome in Navarra-Spain (RESMENA-S) study: a multidisciplinary strategy based on chrononutrition and nutritional education, together with dietetic and psychological control. Nutr Hosp 26, 16-26 (2011).