ARTÍCULO |
Detección de alérgenos de cacahuete mediante un sensor de ADN
Marta Sánchez-Paniagua López1, Gloria Frutos Cabanillas2, M. Jesús Lobo CastaĖón3, Beatriz López-Ruiz1*
1Sec. Dptal. Química Analítica, Facultad de Farmacia, Universidad Complutense de Madrid, 28040, Madrid, EspaĖa. 2Dpto. Estadística e Investigación Operativa, Facultad de Farmacia, Universidad Complutense de Madrid, 28040, Madrid, EspaĖa. 3Dpto. Química Física y Analítica, Facultad de Química, Universidad de Oviedo, 33006, Oviedo, EspaĖa
*e-mail: bealopru@ucm.es
An. Real Acad. Farm. Vol. 80, Nľ 2 (2014), pag. 377-392.
resumen
En el presente trabajo se propone un genosensor electroquímico para la detección de un segmento de ADN que codifica parte de la proteína alergénica Ara h 2 del cacahuete. El genosensor se basa en un ensayo tipo sándwich, el analito hibrida con dos secuencias de bases, una de ellas inmovilizada sobre un electrodo de oro serigrafiado, formando una monocapa autoensamblada. La optimización del dispositivo se realizó utilizando la metodología de Superficies de Respuesta. La máxima respuesta se encontró para concentraciones de sonda de captura y agente bloqueante, 1 mM y 2,5 mM respectivamente. |
Palabras clave: cacahuete; sensor de ADN electroquímico; Superficies de respuesta.
abstract
Detection of peanut allergens by a DNA sensor
In the present work an electrochemical genosensor for detecting a DNA segment encoding part of the allergenic protein peanut Ara h 2 is proposed. Genosensors is based on a sandwich assay format, the analyte hybridized with two base sequences, one immobilized onto a screen printed gold electrode, forming a self-assembled monolayer. The optimization of the device was performed using Response Surface Methodology. The maximum response was found to be 1 ĶM of capture probe concentration and 2,5 mM of blocking agent concentration. |
Keywords: peanut; electrochemical DNA sensor; response surface metodology.
1. INTRODUCcióN
La incidencia de las alergias alimentarias y la gravedad de sus consecuencias han incrementado el interés de los consumidores y ha pasado a ser una cuestión de seguridad alimentaria y salud pública. Se estima que en Europa las alergias alimentarias afectan aproximadamente al 1-2 % de la población adulta y al 5-7 % de la población infantil, aumentando su incidencia en los últimos aĖos. Se hace indispensable tomar medidas para proteger a la población sensible, informando adecuadamente sobre los ingredientes alergénicos que contienen los alimentos e identificando posibles contaminaciones cruzadas. Por ello, se han establecido normas de etiquetado que obligan a indicar su presencia. La legislación alimentaria espaĖola, en sentido estricto, no regula los alérgenos, lo que hace es fijar obligaciones en cuanto a la información en el etiquetado sobre estos ingredientes alérgenos. Existe una lista de 14 grupos de ingredientes alergénicos, entre los que se encuentra el cacahuete y los productos a base de cacahuetes, que es obligado declarar en el etiquetado de los productos alimenticios con independencia del porcentaje en peso que representen en el producto final. Real Decreto 1334/1999, de 31 de julio, por el que se aprueba la Norma general de etiquetado, presentación y publicidad de los productos alimenticios en EspaĖa y Reglamento Nľ 1169/2011 del Parlamento Europeo y del Consejo de 25 de Octubre de 2011 sobre la información alimentaria facilitada al consumidor. Entre esos 14 grupos se encuentra el cacahuete y productos a base de cacahuetes. El cacahuete Arachis hypogaea, conocido también como maní y perteneciente a la familia de las Papilionáceas (leguminosas), es un alimento muy alergénico de gran importancia sanitaria por la incidencia y severidad de la reacción alérgica que produce. En los países desarrollados afecta a un 0,4%-0,6% de los niĖos y un 0,3%-0,7% de los adultos. En EspaĖa, la prevalencia de esta alergia está aumentando, probablemente debido a la mayor popularidad de los productos derivados del cacahuete y a su inclusión en las dietas infantiles.
Algunas de las proteínas de almacenamiento del cacahuete son las responsables de la reacción alérgica. Aproximadamente, el 20% del contenido total proteico del cacahuete se puede atribuir al alérgeno principal Ara h 1 y el 10% al alérgeno Ara h 2. Estos dos alérgenos son la causa del 95% de las reacciones alérgicas de esta legumbre (1). La sensibilización al cacahuete ocurre generalmente durante la infancia y con frecuencia persiste a lo largo de la vida; en este sentido, difiere de otras alergias alimentarias frecuentes (como la alergia a las proteínas de la leche o del huevo), que surgen en la infancia pero generalmente desaparecen en la vida adulta.
Aunque no se conoce con certeza el nivel umbral necesario para causar la reacción alérgica, hay estudios que demuestran que cantidades de proteína del orden de microgramos son suficientes para originar reacciones objetivas en sujetos hipersensibles (2). Por lo tanto, la presencia, no declarada, de trazas de cacahuete en los alimentos puede ser muy peligrosa para estos individuos. Bock y col. realizaron un estudio de 32 casos de muerte por anafilaxia debidas a la ingestión de alimentos alergénicos. Encontraron que más de la mitad de los accidentes mortales (55-67%) se debieron a la ingestión de cacahuete y que en el 90% de los casos, los alérgenos responsables fueron los cacahuetes y las nueces (3). Además de los numerosos productos alimenticios industriales que incluyen cacahuetes como galletas, chocolate, aperitivos, cereales, es habitual encontrarse con alimentos que contienen este alérgeno cuyo origen es la contaminación cruzada producida durante el procesamiento. Por ello, resulta necesario disponer de métodos adecuados de análisis para la detección de alérgenos de cacahuete en productos alimenticios. Se han desarrollado múltiples métodos analíticos basados en la detección de dos tipos de macromoléculas, proteínas y ADN: los primeros detectan alguna de las proteínas alergénicas del cacahuete, mientras que los segundos se basan en la detección de secuencias de oligonucleótidos que codifican alguna de dichas proteínas alergénicas. Los métodos más utilizados para la detección de proteínas son los enzimoinmunoensayos y, en especial, el método ELISA (4-6). Aunque este ensayo ha demostrado ser sensible y versátil, consume tiempo y es relativamente caro, además, los procesos de producción implican un tratamiento térmico que, a menudo, desnaturaliza las proteínas de los alimentos, alterando de este modo su estructura terciaria e impidiendo su detección. Teniendo en cuenta que el ADN no se altera tras someterse a altas temperaturas, los métodos basados en la detección de ADN surgen como una interesante alternativa. Dentro de estos métodos, el primero en aparecer y más utilizado hasta el momento es la reacción en cadena de la polimerasa, PCR, cuantitativa (7-9), método muy sensible que requiere equipos caros y personal cualificado. Estos inconvenientes han impulsado el desarrollo de otros métodos analíticos entre los que se encuentran los biosensores de ADN o genosensores, que presentan ventajas como su menor coste, facilidad de manejo, posibilidad de automatización, etc. En la revisión bibliográfica realizada solo se ha encontrado un genosensor electroquímico aplicado a la detección de alérgenos de cacahuete, basado en la detección del ADN que codifica parte del alérgeno Ara h 1 (10).
En el presente trabajo se propone un genosensor electroquímico para la detección del ADN que codifica parte de la proteína alergénica Ara h 2. Se optó por un formato tipo sándwich en el que todo el analito hidrida con dos secuencias complementarias, una inmovilizada sobre un electrodo de oro serigrafiado y la segunda funcionalizada con biotina. La optimización del dispositivo se llevó a cabo mediante diseĖos de experimentos (DoE), metodología que ha demostrado ser una potente herramienta en química analítica y, especialmente, en la optimización de dispositivos analíticos, aunque su uso no está muy extendido en el campo de los biosensores (11-13).
La metodología de diseĖos de experimentos es una herramienta matemática y estadística cuyos objetivos son seleccionar la estrategia experimental óptima que permita obtener la máxima información con el mínimo coste y evaluar los resultados experimentales obtenidos, garantizando la máxima fiabilidad. Los DoE ofrecen ventajas incuestionables frente a la metodología clásica, entre las que cabe destacar: i) estima el error experimental, ii) calcula la influencia simultánea de los factores, iii) calcula el efecto de las interacciones, iv) con el mismo número de experimentos proporciona mayor información, y v) supone un ahorro de tiempo y esfuerzo así como un menor coste económico (14).
Existen gran variedad de diseĖos de experimentos (15). Una forma de clasificarlos es en función del objetivo analítico propuesto, diferenciándose entre diseĖos de cribado o “screening” y diseĖos de optimización. En los diseĖos screening el objetivo es reducir el posible número de factores experimentales que afectan a la respuesta, descartando los que tienen menor o ninguna influencia en la misma. Estos diseĖos son adecuados en la fase inicial de experimentación y entre los más utilizados se encuentran los DiseĖos Plackett-Burman y DiseĖos Factoriales a dos niveles. Existen dos tipos de diseĖos factoriales, completos y fraccionados. En los diseĖos factoriales completos se utilizan todas las combinaciones posibles de todos los factores a todos los niveles implicados en el experimento, habrá Lk combinaciones de L niveles de k factores. Los diseĖos factoriales fraccionados están formados por una fracción del correspondiente diseĖo factorial completo y contemplan un número menor de experimentos. Una vez realizado el cribado de las variables, se procede a la optimización de la respuesta, para lo cual se utilizan diseĖos factoriales a tres o más niveles, DiseĖos Centrales compuestos, DiseĖos Box-Behnken, DiseĖos Doehlert y DiseĖos D-óptimos entre otros, que aportan información relacionada con el efecto cuantitativo de los factores así como sus interacciones y permiten obtener la Superficie de Respuesta para la variable dependiente en el espacio de las variables independientes.
2. material y métodos
2.1. Reactivos
El 6-mercapto-1-hexanol (MCH), ditiotreitol (DTT), fosfatasa alcalina-estreptavidina (ALP-Strp), 1-naftilfosfato, albúmina humana (BSA), tween 20, las sales para la preparación de las disoluciones tampón (Tris, KCl, MgCl2) y la disolución 20× de fosfato de sodio salino con EDTA pH 7.4 (200 mM fosfato sódico, 3 M NaCl, 20 mM EDTA) fueron suministrados por Sigma-Aldrich (EspaĖa) y utilizados sin purificación previa. El etanol y el ácido sulfúrico fueron adquiridos en Panreac (EspaĖa).
Las disoluciones tampón utilizadas en la preparación del sensor fueron: i) disolucióntampón de inmovilización e hibridación formada por 2×SSPE pH 7,4; ii) disolucióntampón de bloqueo consistente en 5×SSPE pH 7,4 + 5% BSA + 0,1% Tween 20; y iii) disolucióntampón de medida compuesta por 0,5 M Tris-HCl pH 9,8 + 1 mM de MgCl2 + 0,1 M de KCl.
Las secuencias de oligonucleótidos utilizadas fueron suministradas por Sigma-Life Science (Reino Unido). Dichas disoluciones se reconstituyeron en agua Milli-Q y se almacenaron a -20ľC hasta su uso.
2.2. Instrumentación
Las medidas electroquímicas se realizaron en un potenciostato Autolab PGSTAT 12, controlado por el software GPES 4.9005 (EcoChemie B.V, Holanda). Se utilizaron electrodos serigrafiados de oro (referencia 220BT; DropSens, EspaĖa) que constan de una configuración de tres electrodos impresos en una lámina de alúmina, un electrodo de trabajo (diámetro 4 mm) y un electrodo auxiliar, ambos de oro, y un electrodo de pseudo-referencia de plata. La celda electroquímica está limitada por un anillo formado por un polímero aislante que rodea a los tres electrodos y tiene un volumen máximo de 50 mL. Todos los experimentos se llevaron a cabo a temperatura ambiente utilizándose un electrodo para cada medida. Las medidas de pH se realizaron en un pH-metro Crison micropH 2001 (EspaĖa). Para la cuantificación del ADN se utilizó un espectrofotómetro UV-visible Cary 300 Bio (Agilent Tehcnologies, Estados Unidos).
2.3. Procedimiento experimental
2.3.1. DiseĖo del sensor de ADN
Como todos los biosensores, los genosensores o sensores de ADN combinan un elemento de reconocimiento biológico, que en este caso es una secuencia de oligonucleótidos denominada sonda, con un transductor encargado de la medida de la reacción de hibridación entre la sonda y el analito. La sonda utilizada será complementaria a la secuencia de ADN de interés (analito) y formarán un híbrido de Watson-Crick con extrema selectividad incluso en presencia de sondas de oligonucleótidos no complementarias.
La preparación del sensor comienza por la formación de una monocapa autoensamblada (SAM) sobre la superficie del electrodo de oro. Antes de su utilización, los electrodos serigrafiados de oro se sometieron a un proceso de limpieza que consistió en lavado con etanol y agua Milli-Q seguido de un pretratamiento electroquímico de 25 barridos cíclicos entre 0 y +1,6V, a 100 mV/s, en H2SO4 0,1 M, para eliminar los residuos del proceso de fabricación, y así mejorar la sensibilidad y reproducibilidad de los resultados (16).
La monocapa mixta (SAM), consta de una sonda de ADN tiolada y como agente bloqueante, mercaptohexanol (MCH). La inmovilización de la sonda se llevó a cabo mediante un proceso de quimisorción, unión colavente entre los grupos SH de la sonda con el Au del electrodo. El MCH se intercala entre las hebras de ADN quimisorbidas, controlando su disposición sobre el electrodo e impidiendo las adsorciones inespecíficas de la sonda. Las cadenas de ADN se orientan paralelas entre sí y perpendiculares a la superficie electródica (Figura 1), y así, favorecen el proceso de hibridación (17, 18).
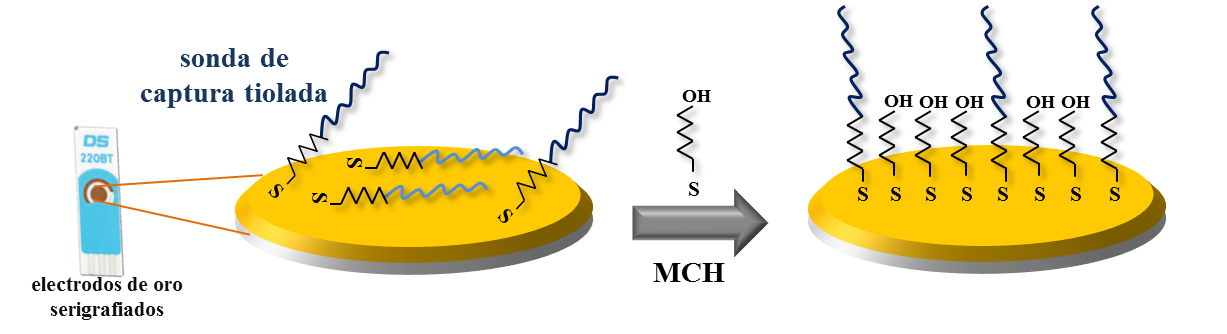
Figura 1.- Formación de una monocapa autoensamblada mixta organizada sobre electrodos de oro serigrafiado.
Para la inmovilización de la sonda de captura se depositaron 15 ĶL de una disolución de la sonda en el tampón de inmovilización, manteniéndose en atmósfera cerrada y saturada con agua durante 19 horas. La monocapa mixta se completó adicionando 10 ĶL de MCH en disolución tampón de inmovilización; dejándose 15 min. Las concentraciones de sonda de captura y MCH cambian y se especifican en cada una de las experiencias.
El genosensor propuesto se basa en un formato tipo sándwich que requiere la hibridación completa del analito con dos sondas diferentes. Una parte de la secuencia del analito hibrida con una sonda indicadora biotinilada (hibridación homogénea, Figura 2.A) y el resto de analito hibrida con la sonda de captura inmovilizada (hibridación heterogénea, Figura 2.B). La hibridación homogénea se llevó a cabo mezclando el analito y la sonda indicadora en la disolución tampón de hibridación, con una concentración de sonda indicadora final de 2 ĶM. La mezcla se calentó a 98ľC para romper las estructuras secundarias de ambas sondas, se sumergió en hielo y se dejó reposar a temperatura ambiente para favorecer la hibridación. Posteriormente, se depositaron 15 ĶL de esta disolución sobre el electrodo modificado, dejando que transcurriese la hibridación heterogénea durante 2 horas a temperatura ambiente.
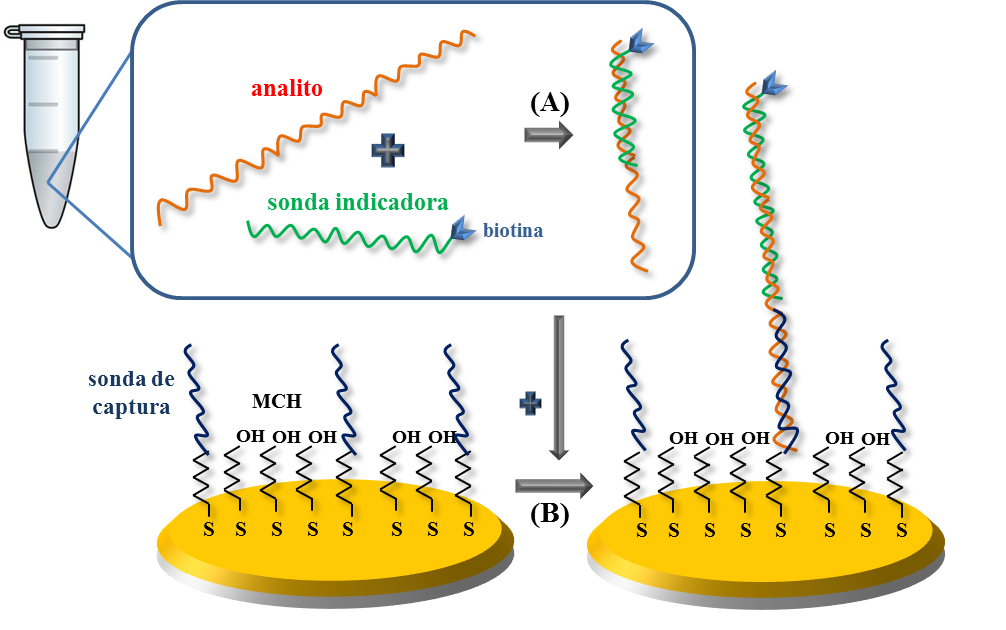
Concluida la etapa de hibridación, se procedió al marcaje enzimático, aĖadiendo 15 ĶL de una disolución del conjugado fosfatasa alcalina-estreptavidina (1,075×10-3 g/L) sobre la monocapa. La sonda indicadora biotinilada se unió al conjugado enzimático por el enlace de alta afinidad estreptavidina-biotina. Finalmente, se llevó a cabo la detección electroquímica para lo cual se aĖadió, a la superficie electródica, 40 ĶL de una disolución de 1-naftilfosfato 4 mM en disolución tampón de medida. El enzima ALP cataliza la conversión de 1-naftilfosfato a 1-naftol, producto electroactivo de estructura plana heterocíclica, cuya oxidación se midió mediante voltamperometría de pulso diferencial (DPV), realizando un barrido de 0 a +0,6 V.
2.3.2. DiseĖo de experimentos
Se realizó un diseĖo factorial completo 32, con 2 variables (concentraciones de sonda de captura y MCH) medidas a 3 niveles. Los resultados se analizaron con el software estadístico Statgraphics® Centurion, versión XVI (19). El modelo que puede soportar el diseĖo utilizado es la función polinómica de segundo grado:
donde los coeficientes b representan los efectos lineales y cuadráticos de las variables X1 (concentración de sonda de captura, M) y X2 (concentración de MCH, M) así como su interacción. La variable dependiente o variable respuesta es la respuesta electroquímica medida por el genosensor (intensidad de corriente, A).
En general, los diseĖos de experimentos se escriben en términos de variables codificadas. En un diseĖo a tres niveles, como es este caso, los niveles de los factores codificados son +1, 0 y -1, donde +1 es el valor máximo considerado, -1 el valor mínimo y 0 el valor central. En la Tabla 1 se muestra la matriz codificada y la matriz experimental y los niveles experimentales fijados para cada uno de los factores estudiados.
Tabla 1. - Matriz codificada y matriz experimental del DiseĖo Factorial 32
Matriz codificada |
Matriz experimental |
||
Niveles del factor 1 |
Niveles del factor 2 |
Concentración de la sonda de captura (ĶM) |
Concentración de MCH (mM) |
0 |
-1 |
1,05 |
0,5 |
0 |
0 |
1,05 |
2,5 |
+1 |
+1 |
2 |
4,5 |
0 |
0 |
1,05 |
2,5 |
0 |
+1 |
1,05 |
4,5 |
-1 |
+1 |
0,1 |
4,5 |
-1 |
-1 |
0,1 |
0,5 |
+1 |
-1 |
2 |
0,5 |
0 |
0 |
1,05 |
2,5 |
0 |
0 |
1,05 |
2,5 |
El diseĖo propuesto se realizó, en el mismo instrumento, el mismo día y por el mismo operador, es decir, en condiciones de repetibilidad. La aleatoriedad en el orden de realización de los experimentos asegura, en la medida de lo posible, que factores incontrolados no afecten de forman sistemática a los resultados obtenidos y se asegura que las estimaciones de la varianza en la repetibilidad reflejen de manera adecuada los aspectos aleatorios del proceso.
3. resultados y discusión
3.1. Selección de las sondas de oligonucleótidos
Para obtener un sensor útil desde el punto de vista analítico, el fragmento de oligonucleótidos elegido como analito tiene que cumplir como mínimo las siguientes características: i) ser específico del cacahuete y ii) no tratarse de una secuencia de ADN muy larga (no más de 100 nucleótidos) para evitar la aparición de estructuras secundarias y apareamientos de bases que dificultarían la hibridación (20).
Como analito se seleccionó una secuencia de 86-mer que codifica parte de la proteína Ara h 2, característica del cacahuete y cuya especificidad se comprobó mediante el programa BLAST (21).
Se seleccionó una sonda de captura de 32 bases tiolada en el extremo 5ę, complementaria en su totalidad con parte del analito y una sonda indicadora de 54 nucleótidos funcionalizada en su extremo 3’ con biotina. La sonda de captura se diseĖó de forma que hibride con la parte del analito que queda libre en la hibridación con la sonda indicadora, formando las tres hebras un dúplex perfecto. De esta forma, se evita que alguna base quede sin hibridar y dé lugar a adsorciones inespecíficas sobre el electrodo y por tanto a errores en la medida.
Para predecir las posibles estructuras secundarias de las sondas y la energía de hibridación del analito con la sonda de captura e indicadora se utilizó el programa informático Mfold web Server (22). La estructura secundaria de la sonda de captura presenta una energía libre DG= -2,9 kcal/mol, la del analito ΔG = -16,71 kcal/mol, la de la sonda indicadora ΔG = -4,39 kcal/mol, el híbrido analito-sonda de captura presenta una energía libre ∆G = -49,8 kcal/mol y el híbrido analito-sonda indicadora ∆G = -85,5 kal/mol. Estos datos demuestran la hibridación espontánea del analito con las dos sondas.
3.2. Selección de las características del diseĖo de experimentos
En la ejecución de un diseĖo de experimentos resulta de gran importancia identificar adecuadamente los factores o variables independientes que pueden afectar a la respuesta (variable dependiente) así como sus niveles experimentales. Una selección errónea de estos niveles podría dar lugar a respuestas muy similares que no se deberían a la no influencia del factor sobre la respuesta sino simplemente a que el factor no afecta en la zona de trabajo, pero puede hacerlo si se amplía dicha zona.
Se comenzó por optimizar la composición de la monocapa, considerando como factores las concentraciones de sonda de captura y de mercaptohexanol. Al seleccionar los niveles, hay que intentar conseguir una SAM homogénea y no demasiado compacta de forma que se permita el contacto entre las hebras complementarias (23).
Experiencias previas demostraron que una concentración de sonda de captura inferior a 0,1 ĶM producía defectos en la monocapa quedando zonas de oro no cubiertas. Por otra parte, se requiere un exceso de sonda de captura en relación al híbrido analito-sonda indicadora con el fin de asegurar que todo el analito incorporado va a poder reaccionar con la sonda de captura para formar el híbrido final. Teniendo esto en cuenta se consideraron como límites experimentales 0,1 y 2 ĶM.
Estudios previos aportan evidencias de que aunque el tratamiento posterior con MCH no permite eliminar completamente el ADN adsorbido inespecíficamente, se logra una disminución considerable de las interacciones inespecíficas y una eficiencia de hibridación superior a la de una SAM pura de ácido nucleico tiolado (24). Con el objetivo de minimizar las absorciones inespecíficas de la sonda de captura sobre la superficie electródica, en todos los casos se aĖadió mayor cantidad de MCH que de sonda de captura. Valores de MCH inferiores a 0,5 mM producían descensos drásticos en la seĖal voltamperométrica del 1-naftol, generándose probablemente una monocapa irregular. Se comprobó que concentraciones superiores a 4,5 mM provocaban el desplazamiento de la sonda de captura. Como consecuencia se seleccionaron como valores límites 0,5 y 4,5 mM.
3.3. Ensayo preliminar
Antes de realizar los diseĖos de experimentos se llevó a cabo un estudio previo con el fin de comprobar que las sondas de captura e indicadora hibridan, de forma selectiva, con el analito y que ese evento de hibridación se traduce en una seĖal electroquímica. Se optó por valores de concentración de sonda de captura y MCH utilizados con éxito en trabajos anteriores (16), 2 ĶM y 4,5 mM respectivamente. El resto de condiciones experimentales fueron las descritas en el Procedimiento Experimental. En la Figura 3 se muestra el voltamperograma obtenido con el analito y con una secuencia no complementaria (nC), ambas a un nivel de concentración 1 nM. El analito dio una seĖal muy superior al blanco mientras que la seĖal generada por la sonda no complementaria fue semejante a la generada por el blanco, lo que demuestra la complementariedad de las sondas con el analito y la especificidad del dispositivo.
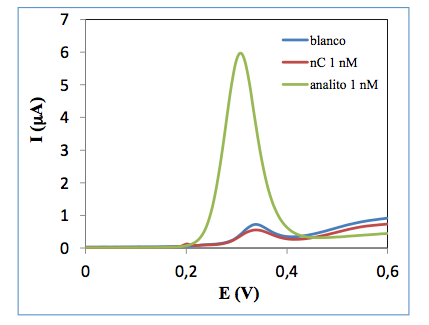
Figura 3.- Voltamperogramas obtenidos con la secuencia analito, una secuencia no complementaria y un blanco. Condiciones experimentales: Voltametría diferencial de impulsos, amplitud de pulso 20 mV, velocidad de barrido 10 mV/s.
3.4. Optimización de la monocapa
Con el objetivo de optimizar la composición de la monocapa se realizaron dos diseĖos factoriales completos 32, ya descritos en el apartado DiseĖo estadístico, siendo las variables independientes las concentraciones de sonda de captura y de MCH. En el primero de ellos se utilizó una concentración de analito de 10 nM. El diagrama de Pareto (Figura 4.A) muestra la influencia de las variables y sus interacciones en la respuesta del sensor, siendo el efecto cuadrático de ambas variables el de mayor influencia en la respuesta. El efecto individual de las variables muestra una influencia menor, siendo positivo el efecto del MCH y negativo el efecto de la sonda de captura. El modelo matemático obtenido corresponde a la función
Sanalito (A) = 5,22737×10-6 + 4,98504×10-6[sonda captura] + 5,97693×10-6[MCH] - 4,50416×10-6[sonda captura]2 + 1,54737×10-6[sonda captura][MCH] - 1,4×10-6[MCH]2
La Figura 4.B muestra los efectos principales sobre la intensidad de corriente producidos por las dos variables. En la Figura 4.C se muestra la superficie de respuesta y en la Figura 4.D las correspondientes curvas de nivel proyectadas en el plano formado por los dos factores. Estas curvas de nivel muestran las condiciones experimentales para las que se obtiene el valor máximo de respuesta así como un espacio próximo al óptimo con la misma respuesta para distintas condiciones experimentales, pudiendo elegir de toda la zona los valores de los factores más convenientes según otros criterios económicos de tipo coste, tiempo, etc. Este máximo viene definido por los valores óptimos de los factores implicados que resultaron ser 1μM de sonda de captura y 2,5 mM de MCH.
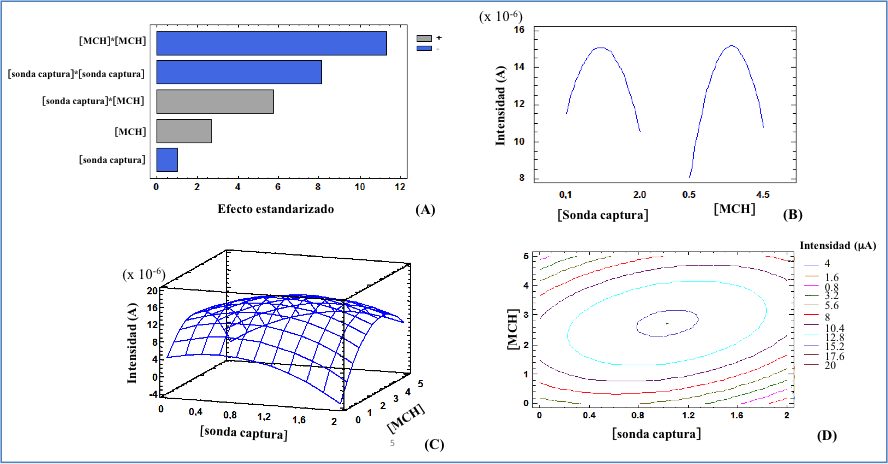
Figura 4.- Resultados del diseĖo factorial 32 con analito 10 nM, (A) diagrama de Pareto estandarizado para intensidad de corriente, (B) gráfica de efectos principales para la intensidad, (C) superficie de respuesta y (D) curvas de nivel.
Debido a la complejidad de la monocapa es habitual la aparición de adsorciones inespecíficas de las sondas y los agentes que la componen y que se traduce en seĖales indeseables. Para averiguar la posible existencia de estas adsorciones y su efecto en la seĖal, se realizó un segundo diseĖo con una disolución blanco. La matriz experimental y los niveles de los factores del diseĖo factorial 32 fueron los mismos que en el diseĖo anterior. Según el diagrama de Pareto (Figura 5.A), el MCH fue el factor de mayor influencia, siendo ésta negativa al igual que la sonda de captura, es decir, el blanco dio menor seĖal al aumentar el contenido de sonda y agente bloqueante. Este hecho se atribuye a la formación, bajo estas condiciones, de una monocapa compacta y sin huecos que dificultan las adsorciones inespecíficas. El modelo matemático ajustado corresponde a la función
Sblanco (A) = 8,43393×10-7 - 1,48473×10-7[sonda captura] - 2,19162×10-7[MCH] - 1,19114×10-8 [sonda captura]2 + 3,32895×10-8 [sonda captura][MCH]+ 2,3625×10-8[MCH]2
La Figura 5.B muestra los efectos principales sobre la intensidad de corriente producidos por las dos variables, no estableciéndose un valor mínimo de respuesta para la sonda de captura en la zona de experimentación. Así mismo, la superficie de respuesta (Figura 5.C) no presenta un óptimo y las curvas de nivel de este ensayo (Figura 5.D) muestran una tendencia que apunta hacia la posibilidad de modificar el campo experimental hacia valores mayores de sonda de captura, sin embargo, mayores concentraciones de sonda de captura dificultarían la reacción de hibridación por impedimento estérico.
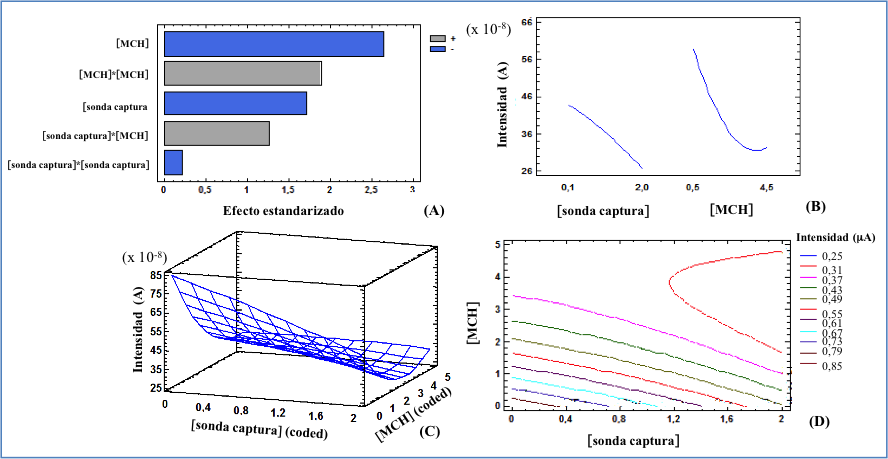
Figura 5.- Resultados del diseĖo factorial 32 con un blanco (A) diagrama de Pareto estandarizado para intensidad de corriente, (B) gráfica de efectos principales para la intensidad, (C) superficie de respuesta y (D) curvas de nivel.
Con el objetivo de conocer la seĖal debida exclusivamente al analito, se generó una nueva variable diferencia (seĖal del analito – seĖal del blanco). El modelo matemático obtenido corresponde a la ecuación
Sanalito-Sblanco (A) =4,39816×10-6 + 5,13019×10-6 [sonda captura] + 6,17592×10-6 [MCH] - 4,49307×10-6 [sonda captura]2 + 1,51579×10-6 [sonda captura][MCH] - 1,42×10-6 [MCH]2
En la Figura 6 se muestran la superficie de respuesta para el analito, el blanco y la diferencia y la curva de nivel para la diferencia en la Figura 6.D. Los valores óptimos prácticamente coincidieron con los obtenidos previamente, 1μM de sonda de captura y 2,5 mM de MCH, ya que la seĖal obtenida por el blanco, en estas condiciones, es prácticamente despreciable frente a la obtenida con el analito. Reducir los valores del blanco es uno de los principales objetivos en estos dispositivos analíticos, para así aumentar la relación seĖal/blanco y, como consiguiente, el límite de detección. Los blancos obtenidos en estas condiciones experimentales hacen que la relación S/B aumente de manera apreciable.

Figura 6.- Curvas de superficie de respuesta: (A) 10 nM de analito, (B) blanco y (C) diferencia 10 nM analito-blanco. (D) Curva de nivel para la variable diferencia.
3.5. Curva de calibrado y estudio de reproducibilidad
Se realizó una curva de calibrado, bajo las condiciones experimentales óptimas obtenidas de los diseĖos de experimentos, 1ĶM de sonda de captura y 2,5 mM de MCH. Se observó una relación lineal entre la intensidad de corriente y la concentración de analito, con un intervalo lineal de 1×10-10 a 1 10-7 M (Figura7), una ecuación I (A) = 2×10-6 C (nM) + 4×10-6, y un coeficiente de regresión: r=0,998. El límite de detección resultó ser 50 pM. Para la reproducibilidad se midió la respuesta de una disolución de analito 10 nM en ocho electrodos diferentes, el coeficiente de variación obtenido fue 7,56 % (n = 8).
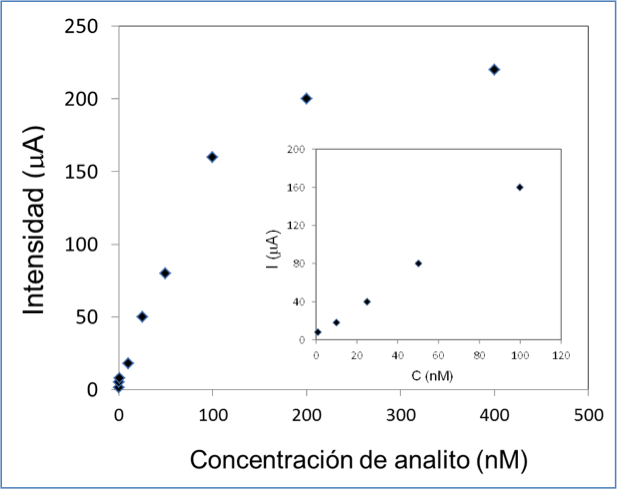
Figura 7.- Curva de calibrado obtenida bajo las condiciones óptimas: 1ĶM de sonda de captura, 2,5 mM de MCH, 2 ĶM de sonda de indicadora, 1,075×10-3 g/L conjugado ALP-Strp y 4 mM naftilfosfato. Inlet: Intervalo lineal.
4. Conclusiones
Las sondas indicadora y de captura seleccionadas hibridan de manera selectiva con el analito, y el sensor de ADN electroquímico resultante permitió la determinación selectiva de una secuencia específica de Ara h 2, proteína alergénica del cacahuete. Mediante el uso de diseĖos experimentales factoriales 32 se obtuvieron superficies de respuesta a partir de las cuales se calcularon los valores óptimos de los factores considerados en la optimización de la monocapa autoensamblada, 1ĶM de sonda de captura y 2,5 mM de mercaptohexanol. El genosensor propuesto presenta una relación lineal entre la respuesta analítica y la concentración de analito en un amplio intervalo concentraciones, entre 0,1 nM y 0,1 μM, con un límite de detección del orden de picomolar y una reproducibilidad satisfactoria, que lo hace idóneo para su utilización como detector del alérgeno Ara h 2.
5. agradecimientos
Los autores agradecen el apoyo financiero obtenido a través del Proyecto Nacional financiado por el Ministerio de Economía y Competitividad. Código: MINECO-13-CTQ2012-31157 y del Grupo de Investigación Universidad Complutense (Ref. Grupo: 950247).
6. referencias
1. Hefle, S. Methods for detecting peanuts in food. Detecting allergens in food; Koppelman, S.J., Hefle, S.L. Ed.; Woodhead Publising; 2006; p 185-186.
2. Morisset, M.; Moneret-Vautrin, D A.; Kanny, G.; Guenard, L.; Beaudouin, E.; Flabbee, J.; Hatahet, R. Thresholds of clinical reactivity to milk, peanut, and sesame in immunoglobulin E-dependent allergies: evaluation by double-blind or single-blind placebo-controlled oral challenges. Clin. Exp. Allergy 33, 1046–1051 (2003).
3. Bock, SA.; MuĖoz-Furlong, A.; Sampson, HA. Fatalities due to anaphylactic reactions to foods. J. Allergy Clin. Immun. 107, 191-193 (2001).
4. Pomés, A.; Helm, RM. ; Bannon, GA. ; Burks, AW. ; Tsay, A.; Chapman, MD. Monitoring peanut allergen in food products by measuring Ara h 1. J. Allergy Clin. Immun. 111, 640-645 (2003).
5. Stephan, O.; Vieths, S. Development of a Real-Time PCR and a Sandwich ELISA for Detection of Potentially Allergenic Trace Amounts of Peanut (Arachis hypogaea) in Processed Foods. J. Agr. Food Chem. 52, 3754–3760 (2004).
6. Zeleny, R.; Schimmel, H. Towards comparability of ELISA results for peanut proteins in food: A feasibility study. Food Chem. 123, 1343-1351 (2010).
7. Hird, H.; Lloyd, J.; Goodier, R.; Brown, J.; Reece, P. Detection of peanut using real-time polymerase chain reaction. Eur. Food Res. Technol. 217, 265-268 (2003).
8. Watanabe, T.; Akiyama, H.; Maleki, S.; Yamakawa, H.; Iijima, K.; Yamazaki, F. A specific qualitative detection method for peanut (Arachis Hypogaea) in foods using polymerase chain reaction. J. Food Biochem. 30, 215-233 (2006).
9. Lopez-Calleja, IM.; de la Cruz, S.; Pegels, N.; Gonzalez, I.; Garcia, T.; Martin, R. Development of a real time PCR assay for detection of allergenic trace amounts of peanut (Arachis hypogaea) in processed foods. Food Control 30, 480-490 (2012).
10. Sun, X.; Guan, L.; Shan, X.; Zhang, Y.; Li, Z. Electrochemical detection of peanut allergen Ara h 1 using a sensitive ADN biosensor based on stem-loop probe. J. Agr. Food Chem. 60, 10979-10984 (2012).
11. Rubio Retama, J.; Sanchez-Paniagua, M.; Hervas Perez, JP.; Frutos Cabanillas, G.; Lopez-Cabarcos, E.; Lopez-Ruiz, B. Biosensors based on acrylic microgels. A comparative study of immobilized glucose oxidase and tyrosinase. Biosens. Bioelectron. 20, 2268-2275 (2005).
12. Alonso-Lomillo, M.A.; Dominguez-Renedo, O.; Ferreira-Goncalves, L.; Arcos-Martinez, M.J. Sensitive enzyme-biosensor based on screen-printed electrodes for Ochratoxin A. Biosens. Bioelectron. 25, 1333-1337 (2010)
13. Venturini Uliana, C.; Olimpio Tognolli, J.; Yamanaka, H. Application of Factorial Design Experiments to the Development of a Disposable Amperometric ADN Biosensor. Electroanal. 23,2607–2615 (2011).
14. Montgomery, D.C; Design and Analysis of Experiments; John Wiley & Sons, United States, 2009.
15. Hibbert, DB. Experimental design in chromatography: a tutorial review. J. Chromatogr. B 910, 2-13 (2012).
16. Miranda-Castro, R.; Lobo-CastaĖón., MJ.; Miranda-Ordieres, AJ.; TuĖón-Blanco, P. Stem-Loop ADN probes for the voltammetric determination of Legionella pneumophila on disposable screen-printed gold electrodes. Electroanal. 21, 267-273 (2009).
17. Herne, TM.; Tarlov. MJ. Characterization of ADN probes immobilized on gold surfaces. J. Am. Chem. Soc. 119, 8916-8920 (1997)
18. Pividori, MI.; Merkoci, A.; Alegret, S. Electrochemical genosensor design: immobiliation of oligonucleotides onto transducer surfaces and detection methods. Biosens. Bioelectron. 15, 291-303 (2000).
19. Statgraphics Centurion. STATGRAPHICS®. Version XVI. 2012. StatPoint Technologies, Inc. www.STATGRAPHICS.com
20. Del Giallo, M.L.; Lucarelli, F.; Cosulich, E.; Pistarino, E.; Santamaria, B.; Marrazza, G.; Mascini, M. Steric Factors Controlling the Surface Hybridization of PCR Amplified Sequences. Anal. Chem. 77, 6324-6330 (2005).
21. Basic Local Alignment Search Tool; www.ncbi.nlm.nih.gov/BLAST
22. Zuker, M. Mfold web server for nucleic acid folding and hybridization prediction. Nucleic Acids Res. 31, 3406-3415 (2003).
23. Levicky, R; Herne, T.M.; Tarlov, M.; Satija, S.K. Using Self-assembly to control the structure of ADN monolayers on gold: a neutron reflectivity study. J. Am. Chem. Soc. 120, 9787-9792 (1998).
24. Lao, R.; Song, S.; Wu, H.; Wang, L.; Zhang, Z.; He, L.; Fan, C. Electrochemical interrogation of DNA monolayers on gold surfaces. Anal. Chem. 77, 6475-6480 (2005).