ARTÍCULO |
Descubrimiento de nuevos antimaláricos a partir de fármacos conocidos mediante cribado in silico e in vitro
Yanetsy Machado Tugores1,5*, Alfredo Meneses Marcel1,5, Yovani Marrero Ponce2,3, Vicente J. Aran4, José Antonio Escario García-Trevijano5, Huong Le Thi Thu2, Rory N. García Sánchez6 y Alicia Gómez Barrio5.
1Departamento de Parasitología, Centro de Bioactivos Químicos, Universidad Central “Martha Abreu” de Las Villas (UCLV), Santa Clara, 54830, Villa Clara, Cuba. 2Unit of Computer-Aided Molecular “Biosilico” Discovery and Bioinformatic Research (CAMD-BIR Unit), Facultad de Química y Farmacia. UCLV, Santa Clara, 54830, Villa Clara, Cuba. 3Institut Universitari de CiŹncia Molecular, Universitat de ValŹncia, Edifici d'Instituts de Paterna, P.O. Box 22085, E-46071, Valencia, EspaĖa. 4Instituto de Química Médica, CSIC, c/ Juan de la Cierva 3, 28006-Madrid, EspaĖa. 5Departamento de Parasitología. Facultad de Farmacia. Universidad Complutense de Madrid. EspaĖa. 6Laboratorio de Investigación de Productos Naturales Antiparasitarios de la Amazonía.
e-mail: ymachadotugores@yahoo.com
Recibido el 12 de julio de 2012
An. R. Acad. Farm. 78, 4, 2012, 461-498
RESUMEN
Existe una urgente necesidad de descubrir nuevas alternativas terapéuticas para el tratamiento de la Malaria, dado que los fármacos disponibles en la actualidad muestran una alta toxicidad así como elevados niveles de resistencia. En el presente trabajo se ha diseĖado un protocolo de cribado virtual constituido por diferentes filtros computacionales con el propósito de identificar nuevos núcleos bases antimaláricos a partir de una biblioteca estructuralmente diversa. Este procedimiento retuvo 38 nuevos hit virtuales de los cuales 12 fueron evaluados experimentalmente frente a Plasmodium falciparum, mostrando 3 de ellos actividad antipalúdica y ninguno mostró citotoxicidad inespecífica. Estos compuestos pueden considerarse como nuevos compuestos líderes, dejando una puerta abierta al desarrollo de nuevos antimaláricos. |
Palabras clave: Antimalárico; Cribado virtual; Plasmodium falciparum.
ABSTRACT
Discovery of new antimalarials from commercial drugs by in silico and in vitro screening
Increased efforts in antimalarial drug discovery are urgently needed. This paper applies a virtual screening protocol consisting of different computational filters in order to identify new antimalarial scaffolds from a structurally diverse library. This procedure has retained 38 new virtual hit which 12 were selected for experimental evaluation against Plasmodium falciparum, 3 of them showed significant antimalarial activity. These compounds have diverse chemical structures unrelated to existing antimalarial drugs can therefore be considered as new lead compounds, which leave an open door to the development of new antimalarials. |
Keywords: Antimalarial drug; Virtual screening; Plasmdium falciparum.
1. introducCIÓn
La Malaria es un problema de salud pública en más de 90 países, habitados por un total de 2.400 millones de personas; representando un 40% de la población mundial. La prevalencia se estima en unos 300-500 millones de casos clínicos y la mortalidad de 1-2 millones de personas anuales (1), cifras que superan las de cualquier otra enfermedad transmisible.
Los fármacos de mayor utilización, como cloroquina y la asociación de sulfadoxina y pirimetamina, presentan beneficios limitados y en determinados casos son de eficacia cuestionable debido a fenómenos de resistencia (2). Desafortunadamente, el desarrollo de nuevas terapias es extremadamente lento y sólo un nuevo antimalárico, el Malarone (GlaxoSmithKline, Brentford, UK), ha sido aprobado por la FDA en la última década.
El costo del descubrimiento de nuevos fármacos excede los 750 millones por cada nueva entidad química, por lo que terapéuticas novedosas para enfermedades olvidadas pueden estar fuera del alcance de países del tercer mundo (3). Universidades e instituciones sin ánimo de lucro pueden jugar un papel importante en la búsqueda de nuevas estrategias terapéuticas, y tienen el potencial para crear nuevos paradigmas de descubrimiento de fármacos antipalúdicos que aumenten la efectividad y eficiencia de los métodos tradicionales de experimentación de “prueba y error”.
Los métodos in silico pueden mejorar significativamente el descubrimiento y posterior desarrollo de fármacos. El uso de cribado virtual de bibliotecas químicas ha emergido como un complemento a los costosos ensayos experimentales (4). En la actualidad, se han llevado a cabo tamizajes in vitro de bibliotecas químicas frente a distintos protozoos como Leishmania major (15 000 compuestos) (5) y Plasmodium falciparum (más de 2 millones de compuestos) (6,7); sin embargo, en ninguno de estos trabajos se han realizado cribados in silico previos a la evaluación experimental, que permita priorizar los compuestos a evaluar y con ello reducir el tiempo y el costo de dicho proceso.
Nuestro grupo de investigación tiene una amplia experiencia en el desarrollo racional de fármacos que incluyen trabajos realizados en protozoos, tales como en Trichomonas vaginalis (8), Trypanosoma cruzi (9), y Plasmodium falciparum (10). En este trabajo, se desarrolla el cribado virtual de una base de datos estructuralmente diversa para la selección de compuestos potencialmente antimaláricos, así como la evaluación in vitro frente a Plasmodium falciparum de aquellos identificados como activos en los estudios in silico con el propósito de descubrir nuevos compuestos líderes con actividad antipalúdica.
2. MATERIAL Y MÉTODOS
2.1. Métodos in silico. Cribado virtual de una base de datos comercial integrado por diferentes filtros
Un buen protocolo de cribado virtual necesita ser rápido, preciso, práctico y que permita cribar grandes bases de datos en aras de priorizar un número reducido de compuestos que pueden ser experimentalmente evaluados (Figura 1).
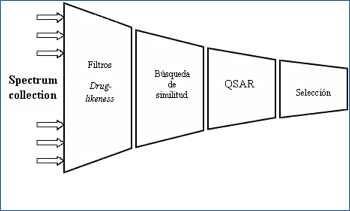
Figura 1.- Esquema del protocolo de cribado virtual “paso a paso” con los diferentes filtros utilizado en este trabajo.
Usando este protocolo, se tamizó la base de datos comercial Spectrum collection (11) (http://www.msdiscovery.com/spectrum.html), compuesta por 2.000 compuestos.
2.1.1. Filtros Drug-likeness
En el desarrollo racional de fármacos, los filtros de cribado virtuales han sido aplicados en todas las etapas del proceso. En las etapas iniciales, se utilizan filtros generales, inespecíficos de la diana farmacológica, para eliminar aquellas estructuras que posean propiedades de no-fármaco (drug-likeness) o ser ligandos de dianas problemáticas (anti-targets). Es decir, consideran si la molécula está dentro de los estándares de relevancia biológica en cuanto a los grupos funcionales que presenta y sus propiedades fisicoquímicas. El método más rápido para evaluar las propiedades drug-likeness de un compuesto es la aplicación de "reglas" cuyos valores asociados se obtienen rápidamente a partir de la estructura utilizando programas computacionales (12).
El filtro ADME (Absorción, Distribución, Metabolismo y Excreción) más clásico es el de Lipinski, basado en 4 propiedades fisicoquímicas del compuesto; pero en la actualidad se ha encontrado que sus márgenes son demasiado estrictos (13-14). Teniendo en cuenta que existían diversos criterios en cuanto a este tipo de reglas, se aplicaron los valores de límites superiores de varios filtros, incluyendo condiciones no incluidas en la regla de Lipinski.
Los descriptores por los cuales realizamos el filtrado de esta base de datos fueron calculados con el programa CDK Descriptor GUI (v0.94) (15) y los datos se almacenaron y procesaron utilizando el programa de Microsoft Excel 2003.
En nuestro caso, un compuesto no fue tomado en consideración si: MW>700g/mol; LogP>7; nHBDon>5; nHBAc>10; nRotB>10; PSA>140 Ā2 (ver Abreviaturas).
La búsqueda de similitud identifica las moléculas de la base de datos que son más similares a los compuestos antimaláricos tomados de referencia, utilizando alguna definición cuantitativa de la similitud estructural intermolecular. Estos métodos entraron en amplio uso desde la década de 1980 y han demostrado ser extremadamente útil en el campo farmacéutico (16-18) ya que son de bajo costo computacional, permitiendo que la búsqueda de grandes bases de datos pueda realizarse rápidamente (19).
Los factores principales que participan en una búsqueda de similitud son los descriptores utilizados y la métrica empleada para establecer la comparación entre pares de moléculas (coeficientes de similitud), permitiendo obtener una lista ordenada en la que, las estructuras más similares a las estructuras de referencia, tienen mayor probabilidad de ser de interés para el usuario (20).
Se utilizaron los descriptores bidimensionales, basados en cadenas de bits de dimensión constante, en las que se indica la ausencia (0) o presencia (1) de una determinada característica en una estructura química, denominados huellas digitales o “fingerprints” y el coeficiente de Tanimoto (Tc) fue utilizado para establecer las métricas de comparación intermolecular (una de referencia vs. una de la base de datos) (14).
Se emplearon como referencia 30 compuestos antimaláricos (querys) con diferentes mecanismos de acción y estructuralmente diversos (Material suplementario 1). De esta manera, cada molécula del Spectrum collection fue ordenada en una posición (ranking) según el coeficiente de similitud respecto a cada una de las estructuras de referencia (14, 15). Los compuestos retenidos son los que se encuentran en la parte superior de la lista con un Tc ≥ 66%.
2.1.3. Estudios de Relación Cuantitativa Estructura-Actividad (QSAR)
Los estudios QSAR constituyen enfoques cuantitativos orientados a encontrar relaciones entre la estructura molecular y las actividades moleculares medidas o calculadas (21). Actualmente, quizás sea el enfoque más utilizado en el diseĖo de fármacos, constando con principios regulatorios de la OECD para la validación de modelos QSAR (22).
Para la obtención de los modelos QSAR en la predicción de actividad antimalárica se confeccionó una base de datos de 2.314 compuestos que se dividieron en 851 y 1.463 activos e inactivos, respectivamente.
Utilizando un análisis de clúster de k-NNCA, implementado en el paquete estadísticoSTATISTICA 6.0. (23, 24), se dividen estos grupos en dos subconjuntos separados, la serie de entrenamiento o calibración (SE) y la serie de predicción o validación (SP). Según muchos autores la “única” condición necesaria y suficiente para poder estimar el valor predictivo de un modelo es comparar los valores predichos y observados de una extensa SP externa.
Se calcularon descriptores moleculares (DMs) implementados en el programa TOMOCOMD-CARDD _ENREF_390(25), de forma que se logra codificar la estructura molecular a través de aplicaciones matemáticas como los índices cuadráticos (IQ)_ENREF_391 (26) lineales (IL) (27) y bilineales (IB) (28, 29).
A partir de los DMs se obtuvieron los modelos o ecuaciones para la identificación de compuestos antimaláricos a través de un análisis discriminante lineal (ADL). Nuestro grupo de investigación ha obtenido modelos usando esta técnica de clasificación para predecir actividad de compuestos con amplios usos farmacológicos, sin embargo las datas utilizadas nunca fueron de esta magnitud (26-30).
Se empleó como criterio de clasificación los valores de diferencia de probabilidades ΔP % (probabilidad con que el modelo clasifica un compuesto como activo menos la probabilidad de clasificar este compuesto como inactivo), de manera que si ΔP %>0, se considera antimalárico. Por el contrario, valores de ΔP %<0, indica la presencia de casos inactivos.
Se han evaluado diferentes parámetros estadísticos para comprobar la calidad y robustez de los modelos obtenidos como la l de Wilks, el valor de F de Fisher (F) y el cuadrado de la distancia de Mahalanobis (D2). De igual forma se calcularon los parámetros: exactitud total (Q), coeficiente de correlación de Mattews (C), sensibilidad (Sens), especificidad (Spec) y razón de falsa alarma (FAR) (29-30).
Para probar la robustez y el poder predictivo de los modelos, no sólo se comparó la predicción obtenida por los modelos con la real de la SP (validación externa); sino que también se llevó a cabo la validación cruzada (VC) dejando un 15 % de los compuestos fuera de la SE generando nuevos modelos de predicción y comprobando el comportamiento de la exactitud del mismo. Este proceder se repite tantas veces hasta que todos los casos son retirados una vez.
El principio de parsimonia (“Occam’s Razor”) fue tomado en cuenta para la selección del número óptimo de variables en cada modelo (31).
2.1.3.1. Sistema multiclasificador ensamblado (SMCs) basado en modelos QSAR.
No existe todavía un clasificador por excelencia; para un problema determinado es difícil seleccionar cual será el clasificador que logre encontrar una mejor frontera de decisión para separar las clases. Por ello, se utilizó un clasificador ensamblado o multiclasificador, a partir de todos los modelos QSAR considerados. Este sistema tiene una tendencia general a mejorar los resultados de las clasificaciones combinando adecuadamente varios clasificadores (en nuestro caso son los modelos individuales) (32, 33).
Una de las condiciones para obtener buenos resultados es lograr la diversidad de los modelos individuales, y para “cuantificar” las correlaciones entre ellos fueron seleccionadas las medidas de diversidad de desacuerdo (D) y de doble fallo (DF) (34). El desacuerdo se basó en aquellos casos que fueron clasificados de manera diferente por dos modelos individuales (se escoge el valor máximo) y el doble fallo tiene en cuenta aquellos casos en que ambos modelos se equivocan en su clasificación (escogiéndose el mínimo valor). De esta forma quedan seleccionados los modelos con mayor diversidad en la información brindada (34, 35).
El método escogido para realizar el SMCs se denomina no entrenado (voto no ponderado) donde se combina, en una matriz, los ΔP de los casos por todos los modelos analizados (expresando la probabilidad de ser activos o inactivos). Mediante la fusión de diferentes funciones matemáticas (media, mediana, valor mínimo y máximo y producto) de todos los ΔP se puede encontrar la mejor combinación de modelos para el cribado de grandes bases de datos (36-38).
2.2. Métodos in vitro
El cultivo de Plasmodium falciparum fue mantenido en medio RPMI 1640 suplementado con Albumax (Gibco) 0,5 %, Hipoxantina (Sigma) 0,1% y glóbulos rojos (grupo O+ o A+, 2% hematocrito). Se incubaron a 37°C y 5% CO2 (39).
2.2.1. Microtest in vitro basado en florescencia para evaluar actividad antimalárica
Usando placas de 96 pocillos se distribuyen 50 Ķl/pocillo de los productos previamente disueltos en dimetilsulfóxido (Panreac), utilizándose 3 réplicas por cada concentración, así como controles con Cloroquina (CQ) (Sigma) y dimetilsulfóxido al 0.2 %. Posteriormente se distribuyeron 50 Ķl/pocillo del cultivo de Plasmodium falciparum de las cepas 3D7 y Dd2, sensible y resistente a CQ, respectivamente, con un hematocrito del 4 % y una parasitemia del 1 %, dejando controles de crecimiento y de glóbulos rojos no parasitados. Después de 48 h de incubación, se aĖaden 100 Ķl/pocillo de SYBR-Green (Applied Biosystem) (0.2 Ķl/ml) en una solución de buffer de lisis y se agitan las placas durante 15 min protegidas de la luz. Seguidamente se mantienen durante una hora en reposo para favorecer la acción del agente intercalante con el DNA parasitario.
La lectura de la fluorescencia se realiza en un espectrofluorímetro (Marca Sunrise RC - TECAN) a una longitud de onda de excitación de 485nm y de emisión de 535nm (40). El porcentaje de inhibición (IC) se calcula usando la fórmula siguiente:

Donde: IF = Intensidad de Fluorescencia
2.2.3. Citotoxicidad inespecífica en macrófagos
Se mide la respiración celular como indicador de viabilidad celular basado en la reducción a nivel mitocondrial de MTT [bromuro de 3-(4,5-dimetiltiazol-2-il)-2,5-difeniltetrazolio] (Sigma) (41). Se emplearon macrófagos de la línea J774 mantenidos en frascos de cultivo celular de 75 cm3, mediante pases sucesivos en medio RPMI-1640 suplementado con 10 % de suero bovino fetal, a 37 °C con 5 % de CO2. Se despegan las células con una solución de EDTA/Tripsina en Fosfato buffer salino (PBS) a 1 g/l. Se centrifugan a 1.500 rpm/5 min y se resuspenden en medio fresco para determinar su concentración y viabilidad a través del recuento en cámara de Neubauer con una solución de azul tripán (Sigma).
El cultivo se ajusta a 70.000 macrófagos en 100 Ķl/pocillo. Después de 24 h de incubación a 37 °C con 5 % de CO2, se retira el medio y se aĖaden 200 Ķl de los productos a evaluar previamente disueltos. Por cada concentración de los productos se utiliza un pocillo sin macrófagos.
También se utilizan controles de dimetilsulfóxido al 0,2 % y de crecimiento de células. Tras 24 h en contacto con los productos se retira el medio y se aĖaden 100 Ķl/pocillo de MTT (0,4 mg/ml) en PBS, incubándose a 37 °C/1 h. Los cristales de formazán se disuelven con 100 Ķl/pocillo de dimetilsulfóxido. Se leen las absorbancias a 595 nm y la actividad citotóxica se calcula empleando la formula:
![]()
Donde: %C = porcentaje de citotoxicidad inespecífica
A= absorbancia.
2.2.4. Test de inhibición de la biomineralización de la ferroprotoporfirina IX (FBIT)
En Plasmodium spp., la eliminación de la ferroprotoporfirina IX (FPIX) durante el proceso de degradación de la hemoglobina, es un mecanismo indispensable para la supervivencia del parásito por lo que un fármaco capaz de inhibir la polimerización de la FPIX, podría ser letal para el parásito (42).
En una placa de 96 pocillos no estéril se colocó 50 ml de solución de clorhidrato de hemina bovina en DMSO por pocillo a una concentración de 0.5 mg/ml, 100 ml de buffer acetato de sodio pH 4.4 por pocillo y 50 ml de cada uno de los compuestos en estudio disueltos en DMSO. Se utilizaron como controles 50 ml de DMSO y 50 ml de cloroquina difosfato (CQ).
Para cada concentración se colocaron 3 replicas, así como para los controles. Las placas se incubaron a 37 °C por 18-24 horas y luego se centrifugaron a 3000 rpm durante 5 minutos eliminando el sobrenadante.
A continuación, se aĖadió 150 ml de DMSO en cada pocillo y se centrifugó nuevamente la placa a 3000 rpm durante 5 minutos, eliminándose de nuevo el sobrenadante.
Posteriormente se colocaron 150 ml de hidróxido de sodio 0,1 N por pocillo, realizándose la lectura de la absorbancia a una longitud de onda de 405 nm en un lector ELISA (42).
El porcentaje de inhibición de cada producto de síntesis fue calculado mediante la siguiente fórmula:
% inh = PAC – PAE x 100 PAC |
% Inh = Porcentaje de Inhibición PAC = Promedio de la absorbancia del control PAE = Promedio de la absorbancia del compuestos de síntesis. |
3. RESULTADOS
3.1. Métodos in silico.
Para cribar grandes bases de datos, se siguió un protocolo de cribado virtual “paso a paso” que permite evaluar de forma jerárquica aquellos compuestos candidatos a ser evaluados experimentalmente. El primer paso consistió en reducir el número de compuestos empleando los filtros de Drug-likeness “optimizando” de alguna manera simultáneamente la potencia y la farmacocinética (43). A partir del filtrado de la base de datos con las reglas ADME empleadas (13-14), se obtuvieron 1.394 compuestos que cumplieron los criterios establecidos.
El siguiente paso reduce de forma rigurosa la biblioteca de compuestos por el análisis de similitud con los compuestos de referencia, obteniéndose una quimioteca más focalizada, pues todos los compuestos incluidos en ella presentan rasgos comunes a los compuestos de referencia. Se retuvieron 154 compuestos con un Tc> 66% (Material suplementario 2).
Se obtuvieron 37 modelos QSAR los cuales mostraron valores de l de Wilks<0,49, Q>86 %, FAR<14,4 %, C>0,71, Sens>80%, Spec>78%, D2>4,50. Estos resultados en los parámetros estadísticos apuntan a una buena calidad para la predicción de actividad antimalárica por parte de los modelos. El modelo 37 mostró el desempeĖo mas óptimo, tanto para la SE como para la SP (Material suplementario 3).
En la figura 2, se ilustran los resultados del ejercicio de VC para el modelo 37 donde se observa que la exactitud de predicción del modelo se mantiene alrededor del 90%; por lo que puede concluirse que el mismo tiene una gran estabilidad a perturbaciones dentro de la SE, por lo tanto se trata de un modelo adecuada calidad.
El filtrado de la base de datos mediante la aplicación de reglas ADME permitió “optimizar” simultáneamente la potencia y la farmacocinética/toxicología (13-14) y el estudio de similitud molecular redujo de forma más rigurosa la biblioteca de compuestos pues los compuestos incluidos en ella presentan rasgos comunes a los querys de referencia. Esto no limita que salgan compuestos con núcleos bases nuevos, pero sí garantiza que aquellos que pasen estos filtros estarán en el dominio de aplicabilidad de los modelos utilizados para la predicción.
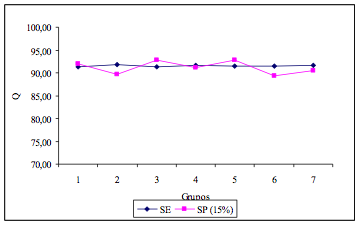
Figura 2.- Resultados de la VC del modelo 37 dejando el 15% fuera de la SE.
Existen modelos publicados por nuestro grupo para la selección de antimaláricos, pero estos carecen de un adecuado dominio de aplicación (10) y no han sido obtenidos con la mezcla de todos los índices (lineales, bilineales y cuadráticos). Nosotros, incluimos en la SE sólo moléculas con IC50<20 ĶM como activas, permitiendo con este punto de corte, identificar sustancias con una potencia adecuada. Por primera vez, se fusionaron los modelos individuales mejorando los parámetros estadísticos de los mismos.
En el problema de la modelación molecular, es típico que exista gran cantidad de casos parecidos con clases diferentes, que hace difícil encontrar los patrones que caracterizan cada una de las clases del problema.
Por esto, surge la idea de usar un sistema que combine varios modelos de clasificadores en un SMC (44). _ENREF_248. Una de las condiciones para obtener buenos clasificadores de base es lograr la diversidad de los mismos (35), por ello se realizó un estudio de diversidad con los 37 modelos obtenidos que nos permitió obtener un menor número de clasificadores a combinar, no tomando en consideración aquellos que coincidieran en la información brindada. En este análisis de diversidad se seleccionaron los 12 modelos que maximizaron el valor promedio del D y minimizaron el valor promedio del DF como medidas de diversidad.
Una vez que se seleccionaron los modelos con mayor diversidad se realizó el análisis del voto no ponderado. Los 12 modelos más diversos se combinaron mediante la fusión de diferentes funciones matemáticas (media, mediana, valor mínimo y máximo y producto de los ΔP). En la Tabla 1 se observa que la combinación de los modelos 2-35-37 superó el resultado del mejor modelo individual en cuanto a la exactitud en la clasificación con un 92, 06% y disminuyó los compuestos inactivos clasificados como activos (FAR).
Tabla 1.- Comparación del modelo individual vs SMC.
SP |
Q |
FAR |
Modelo 37 (mejor modelo individual) |
91,36 |
4,59 |
SMC voto no ponderado (3 modelos) |
92,06 |
3,83 |
Por lo tanto, este ensamble es el que brinda mayor información y será utilizado para la predicción de actividad antimalárica en lugar de utilizar todos los modelos originales o el mejor modelo individual que fue el 37.
Las ecuaciones que corresponden a los modelos seleccionados en el SMC, así como la clasificación expresada en probabilidades, se pueden ver en el Material suplementario 4 y 5.
Los 154 compuestos retenidos del estudio de similitud molecular fueron cribados por los modelos 2-35-37, siendo clasificados como activos 38 compuestos (Material suplementario 6).
El protocolo del cribado virtual utilizado, permitió reducir la cantidad de compuestos gradualmente después de cada filtro de forma rápida y precisa para ser evaluados experimentalmente. Spectrum collection está aprobada por la FDA y es de uso clínico por lo que la aplicación en humanos ya ha sido evaluada, existiendo en la mayoría de los casos datos de la farmacocinética y toxicidad (11); además pueden ser adquiridos por la compra directa a sus propietarios, evitándose la síntesis o aislamiento.
Otro aspecto crucial es que la base de datos cubre un espectro amplio de actividades y estructuras, siendo favorable para nuestro propósito de encontrar nuevos núcleos bases y si es posible nuevos mecanismos de acción, dado que los modelos QSAR no fueron entrenados con series congenéricas, ni estructurales, ni de mecanismo de acción.
El esquema de trabajo utilizado permite la retroalimentación de los resultados obtenidos ya que demostrando ser factible, se puede aumentar la base de datos con compuestos corroborados experimentalmente por nuestro equipo de trabajo, así como modificar los criterios de inclusión según nuestros intereses.
3.2. Métodos in vitro
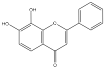
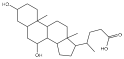
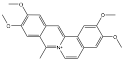
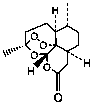
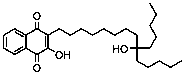
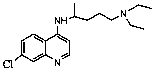
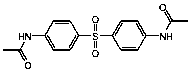
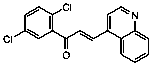
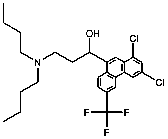
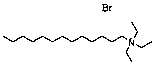
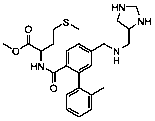
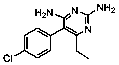
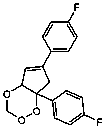
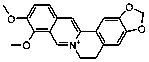
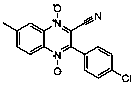
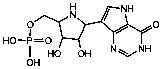
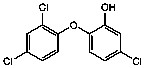
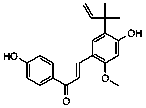
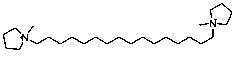
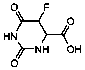
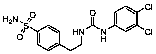
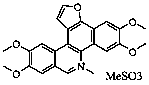
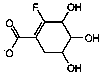
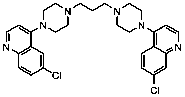
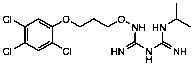
Se seleccionaron 12 productos considerando la diversidad estructural de los mismos y la disponibilidad en Sigma-Aldrich y se evaluaron frente a las cepas 3d7 y Dd2 de Plasmodium falciparum. Los resultados de los 3 compuestos que mostraron actividad relevante se reflejan en la Tabla 2.
Tabla 2.- Clasificación y corroboración in vitro frente a Plasmodium falciparum
Compuesto |
Actividad reportada |
∆P Modelos |
IC50Ķg/ml (ĶM) |
|||||
No. |
Estructura química |
2 |
35 |
37 |
3D7 |
Dd2 |
||
4 |
|
Protector vascular, antihemorrágico |
94,9 |
16,4 |
72,5 |
3,3 (13,2) |
3,1 (12,0) |
|
8 |
|
Anticolelitogénico |
92,5 |
68,5 |
63,4 |
6,2 (15,8) |
4,1 (10,4) |
|
11 |
|
Citostático, agente intercalante |
95,3 |
93,3 |
92,8 |
(0,31) |
(0,33) |
|
Leyenda: No. 4= 7,8 Dihidroxiflavona (7,8-Dihydroxyflavone Hydrate).
No. 8= Ursodiol (Ursodeoxycholic acid).
No.11= Clorhidrato de Coralina (Coralyne Chloride Hydrate).
Entre paréntesis se muestra la IC50 en ĶM.
En el ensayo de citotoxicidad inespecífica frente a cultivos de macrófagos se obtuvo como resultado que los compuestos activos frente a Plasmodium falciparum no mostraron actividad citotóxica a la máxima concentración ensayada (50 ĶM) por lo que se clasifican como no tóxicos.
De los 3 compuestos evaluados en el ensayo de FBIT, solo el compuesto 11 mostró inhibición de la biomineralización de la β-hematina con una IC50 =28 ĶM, siendo esta concentración similar a la reportada para la CQ en esta prueba (45); sugiriendo que el mecanismo por el cual actúa frente a Plasmodium falciparum puede estar relacionado con el proceso de la degradación de la hemoglobina.
Ninguno de los 3 compuestos que mostraron actividad in vitro frente a Plasmodium falciparum tenían acción frente a este parásito en reportes previos; sin embargo, el compuesto 4 ha sido evaluado frente a Trypanosoma brucei (46). Para el compuesto 8 no se ha descrito previamente su actividad frente a Plasmodium spp, sin embargo se ha descrito su actividad inhibitoria frente a una enzima alanina aminopeptidasa de la familia M1 de Plasmodium falciparum (M1AAP) mediante un ensayo que midió la hidrólisis de un sustrato péptico fluorogénico (H-Leu-NHMec).
La enzima es una aminopeptidasa citosólica M1AAP relacionada con la etapa terminal del proceso de digestión de la hemoglobina en el eritrocito por lo que puede ser una diana importante para la quimioterapia de la enfermedad (47).
El compuesto 11 muestra semejanza en cuanto a actividad citostática con la berberina, conocido antimalárico que actúa sobre el ADN (48). También tiene una estructura similar a otras benzo[c]fenantridinas cuaternarias las cuales son efectivas inhibidores de topoisomerasas humanas; sin embargo estas no han mostrado significativa actividad in vitro frente a Plasmodium falciparum (49).
Por todo ello se ha sugerido que el modo de acción frente al parásito para este tipo de compuesto puede ser diferente al efecto en las células cancerosas humanas.
4. AGRADECiMIENTOS
Los autores agradecen el apoyo financiero del Proyecto PCI-AECI (A/030156/10) titulado “Selección racional, obtención y evaluación de nuevos compuestos antiprotozoarios: antitrichomonas y antimaláricos” al igual que a la “Comisión Interministerial de Ciencia y Tecnología” (CICYT) espaĖola (referencia del Proyecto: SAF2009-10399). Uno de los presentes autores (Dr. Yovani Marrero Ponce) agradece al programa de ‘Estades Temporals per a Investigadors Convidats’ por trabajar en la Universidad de Valencia (2011) y el Dr. Alfredo Meneses Marcel agradece al programa de visitantes distinguidos e investigadores extranjeros en la UCM (Grupo Santander 2010).
5. REFERENCIAS
1. Snow, R.W.; Guerra, C.A.; Noor, A.M.; Myint, H.Y.; Hay, S.I.; Nature 2005, 434, 214–217.
2. González, P. D.; Enfermedades Emergentes, 2005, 7(1), 40-43.
3. Rosenthal, P. J.; Journal of Experimental Biology, 2003, 206:3735–3744.
4. Seifert, M.; Wolf, K.; Vitt, D.; Biosilic, 2003, 1(4), 143-9.
5. George, S.; Bishop, J. V.; Titus, R. G.; Selitrennikoff, C. P.; Antimicrobial Agents and Chemotherapy, 2006, 50(2), 474–479.
6. Chong, C. R.; Lirong Shi, X.; Liu, J. O.; Nature chemical biology, 2006, 2, 415-416.
7. Gamo, F. J.; Sanz, L. M.; Vidal, J.; Cozar, C.; Alvarez, E.; Lavandera, J. L.; Vanderwall, Green, D. V. S.; Kumar, V.; Hasan, S.; Brown, J. R.; Peishoff, C. E.; Cardon, L. R.; Garcia-Bustos, J. F.; Nature, 2010, 465(7296), 305-10.
8. Meneses-Marcel, A.; Marrero-Ponce, Y.; Machado-Tugores, Y.; Montero-Torres, A.; Montero Pereira, D.; Escario, J. A.; Nogal-Ruiz, J. J.; Ochoa, C.; Arán, V. J.; Martínez-Fernández, A. R.; García Sánchez, R. N.; Bioorganic Medical Chemestry Letter, 2005, 17, 3838-3843.
9. Montero-Torres, A.; Vega, C. A.; Marrero-Ponce, Y.; Rolón, M.; Gómez-Barrio, A.; Escario, J. A.; Arán, V. J.; Martínez-Fernández, A. R.; Meneses-Marcel, A.; Bioorganic & Medical Chemestry, 2005, 13, 6264-6275.
10. Marrero-Ponce, Y.; Montero-Torres, A.; Romero-Zaldivar, C.; Iyarreta-Veitía, I.; Mayón Peréz, M.; García Sánchez, R.; Bioorganic & Medical Chemestry, 2005, 13, 1293-1304.
11. MicroSource Spectrum collection (http://www.msdiscovery.com/spectrum.html)
12. Schneider, G.; Baringhaus, K.-H.; Molecular Design: Concept and Applications; Weinheim: Wiley-VCH Verlag GmbH & Co. KGaA, 2008.
13. Lipinski, C.; Hopkins, A.; Nature, 2004, 432(7019), 855-861.
14. Oprea, Tudor I.; Journal of Computer-Aided Molecular Design, 2000, 14(3), 251-264.
15. Guha, R.; [http://www.rguha.net/code/java/cdkdesc.html]: Boston, MA, USA, 1991.
16. Downs, G. M.; Willett, P.; Rev. Comput. Chem. 1995, 7, 1.
17. Willett, P.; Barnard, J. M.; Downs, G. M. J. Chem. Inf. Comput. Sci. 1998, 38, 983.
18. Bajorath, J.; J. Chem. Inf. Comput. Sci. 2001, 41, 233.
19. Sheridan, R. P.; Kearsley, S. K.; Drug Discov Today 2002, 7, 903.
20. Hert, J.; Willett, P.; Wilton, D. J.; J. Chem. Inf. Comput. Sci. 2004, 44, 1177.
22. OECD. In Guidance Document on the Validation of (Quantitative) Structure-Activity Relationship [(Q)SAR] Models, Series on Testing and Assessment. Paris, 2007, 69, 154.
25. Marrero-Ponce, Y.; Romero, V.; TOMOCOMD software. Central University of Las Villas; 2002. TOMOCOMD (TOpological MOlecular COMputer Design) for Windows, version 1.0 is a preliminary experimental version; in future a professional version can be obtained upon request to Y. Marrero: yovanimp@qf.uclv.edu.cu or ymarrero77@yahoo.es.
26. Marrero-Ponce, Y.; Bioorganic & Medical Chemestry, 2004, 12, 6351-6369.
27. Marrero-Ponce, Y.; Castillo-Garit, J. A.; Olazabal, E.; Serrano, H. S.; Morales, A.; Castanedo, N.; Ibarra-Velarde, F.; Huesca-Guillen, A.; Sanchez, A. M.; Torrens, F.; Castro, E. A.; Bioorganic & Medical Chemestry, 2005, 13, 1005-1020.
28. Marrero-Ponce, Y.; Journal of Chemical Information and Computer Sciences, 2004, 44, 2010-2026.
31. Baldi, P.; Brunak, S.; Chauvin, Y.; Nielsen, H.; Bioinformatics, 2000, 16(5), 412-424.
32. Opitz, D.; Maclin, R. Journal of Artificial Intelligence Research 1999, 11, 169.
33. Goebel, K.; Yan, W.; Seventh International Conference in Information Fusion, 2004.
34. Kuncheva, L. I.; Combining Pattern Classifiers. Methods and Algorithms; Wiley Interscience, 2004.
36. Marrero-Ponce, Y.; Khan, M. T.; CasaĖola Martin, G. M.; Ather, A.; Sultankhodzhaev, M. N.; Torrens, F.; Rotondo, R.; ChemMedChem, 2007, 4(2), 449.
37. CasaĖola-Martin, G. M.; Marrero-Ponce, Y.; Khan, M. T. H.; Ather, A.; Sultan, S.; Torrens, F.; Rotondo, R.; Bioorganic & Medical Chemestry, 2007, 15(3), 1483-1503.
38. Bauer, E.; Kohavi, R.; Machine Learning, 1999, 36, 105.
39. Trager, W.; Jensen, J. B.; Science, 1976, 193, 673-675.
41. Hattori, Y.; Nkanishi, N.; Cellular Inmunology, 1995, 165, 7-11.
42. Deharo, E.; García, R.N.; Oporto, P.; Gimenez, A.; Sauvain, M.; Jullian, V.; Ginsburg, H.; Exp. Parasitol. 2002, 100, 252–256.
43. Oprea, T. I.; Matter, H.; Current Opinion in Chemical Biology, 2004, 8, 349-358.
44. Dietterich, T. G.; Ensemble methods in machine learning, In Multiple Classifier Systems; Springer-Verlag: Berlin, 2000; p 1-15.
45. PubChem. BioActivity Services. In vitro inhibitory concentration against ferriprotoporphyrin in biomineralisation assay (FBIT). Retrieved January 19, 2011, http://pubchem.ncbi.nlm.nih.gov/assay/assay.cgi?cid=2719
46. Mackey, Z. B.; Baca, A. M.; Mallari, J. P.; Apsel, B.; Shelat, A.; Hansell, E. J.; Chiang, P. K.; Wolff, B.; Guy, K. R.; Williams, J.; McKerrow, J. H.; Chemical Biology & Drug Design, 2006, 67: 355–363.
47. PubChem. BioActivity Services. Inhibitors of Plasmodium falciparum M1- Family Alanyl Aminopeptidase (M1AAP). Retrieved January 12, 2011, from
48. http://0-pubchem.ncbi.nlm.nih.gov.opac.acc.msmc.edu/assay/assay.cgi?q=da&cid=72
49. Mazzini, S.; Bellucci, M. C.; Mondelli, R.; Bioorganic & Medicinal Chemistry, 2003, 11(4), 505-514
50. Nyangulu, J. M.; Hargreaves, S. L.; Sharples, S. L.; Mackay, S. P.; Waigh, R. P.; Duval, O.; Mberu, E. K.; Watkins, W. M.; Bioorganic & Medicinal Chemistry Letters, 2005, 15, 2007–2010.
6. Abreviaturas
ADN |
Ácido desoxirribonucleico |
CARDD |
Computer-Aided Rational Drug Design |
D |
Desacuerdo |
DF |
Doble fallo |
FDA |
Agencia de Alimentos y Medicamentos del gobierno de los Estados Unidos |
IB |
Índices bilineales |
IC50 |
Concentración Inhibitoria 50 |
IL |
Índices lineales |
IQ |
índices cuadráticos |
K |
Electronegatividad en la escala de Mulliken |
k-MCA |
Análisis de Clúster de k-Medias |
k-NNCA |
Algoritmo de Clúster de k-Vecinos más Próximos |
LogP |
Coeficiente de partición octanol/agua |
MTT |
3-(4,5-dimethylthiazole-2-yl)-2,5 diphenyl-tetrazolium bromide |
nHBAc |
Número de aceptores de puente de hidrógeno |
nHBDon |
Número de dadores de puente de hidrógeno |
nRotB |
Número de enlaces rotables |
PBS |
Fosfato buffer salino |
PSA |
Área superficie polar |
QSAR |
Quantitative Structure Activity Relationships |
Sens |
Sensibilidad |
Spec |
Especificidad |
TOMOCOMD |
TOpological MOlecular COMputational Design |