ARTÍCULO |
Aplicación de la topología molecular en la búsqueda de nuevos compuestos derivados del 4-nitro-imidazol activos frente al Tripanosoma brucei
Boris Guzmán Fernández, Riccardo Zanni, Marina Pellicer, Maria Galvez-Llompart, Ramón Garcia-Domenech *.
Departamento de Química Física, Facultad de Farmacia, Universitat de Valencia. Avda. V. A. Estelles, s/n, 46100-Burjassot, Valencia, Spain.
e-mail: ramon.garcia@uv.es
Recibido el 10 de octubre de 2012
An. R. Acad. Farm. 78, 4, 2012, 511-526
RESUMEN
La Tripanosomiasis Humana Africana, causada por el parásito protozoario de la especie Trypanosoma brucei, está caracterizada por un proceso infectivo crónico discapacitante que afecta a millones de personas en todo el mundo. El arsenal terapéutico frente a esta enfermedad, requiere generalmente suministración por vía parenteral, lo que dificulta la adhesión y accesibilidad del paciente al tratamiento. Se ha desarrollado un modelo topológico-matemático encaminado a buscar nuevos compuestos derivados del 1-aril-4-nitro-1H-imidazol con potencial actividad anti-tripanosómica. Utilizando el análisis lineal discriminante se ha obtenido un modelo capaz de predecir correctamente la actividad del 93% de los compuestos estudiados. Se ha sometido al modelo a una validación interna por medio del test de Jack-knife y de una validación cruzada. Tras realizar un cribado molecular virtual se proponen diez nuevos derivados imidazólicos con potencial actividad anti-tripanosómica. |
Palabras clave: Trypanosoma brucei; Análisis QSAR; Topología molecular.
ABSTRACT
Molecular topology application in the search for new active compounds derived from 4-nitro-imidazol against Trypanosoma brucei
Human African Trypanosomiasis, caused by the protozoan parasite Trypanosoma brucei, is characterized by a disabling chronic infectious process affecting millions of people worldwide. The therapeutic arsenal against this disease usually requires intravenous suministración, hindering accessibility and adherence to therapy. It has developed a topological mathematical model aimed to finding new compounds derived from 1-aryl-4-nitro-1H-imidazol with potential anti-trypanosome activity. Using linear discriminant analysis (LDA) was obtained a model capable of predicting correctly the activity of 93% of the studied compounds. The model has been subjected to an internal validation using the jack-knife test or leave-one-out and an internal cross-validation. Following a virtual sweep or virtual screening ten new imidazole derivatives are proposed, with potential anti-trypanosome activity. |
Keywords: Trypanosoma brucei; QSAR analysis; Molecular Topology.
1. introducCIÓn
La Tripanosomiasis Humana Africana (THA) está causada por el parásito protozoario de la especie Trypanosoma brucei sensu lato, y pertenece a las enfermedades tropicales desatendidas, un grupo de infecciones crónicas discapacitantes que afectan a más de un millón de personas en todo el mundo. África es el continente más afectado por esta enfermedad, siendo las zonas rurales, urbanas pobres o zonas en conflicto donde mayor presencia tiene (1). Se calcula que tiene un impacto económico estimado de más de 1.300 millones de dólares por aĖo en pérdidas en cuanto a recursos para los países en vías de desarrollo.
La transmisión de la tripanosomiasis animal se produce principalmente a través de su vector natural, la mosca tse-tsé, aunque también puede darse la transmisión mecánica por picadura de otros insectos.
La THA (o enfermedad del sueĖo) es causada por el Trypanosoma brucei gambiense o bien Trypanosoma brucei rhodesiense; se calcula que se dan más de 10.000 casos al aĖo de infección en humanos. T. b. gambiense, presente comúnmente en África occidental y central, es la causa del 90% de los casos reportados de THA. Se desarrolla como una enfermedad crónica que se caracteriza por una progresión lenta. En la etapa tardía suele presentarse un cuadro de encefalitis que puede derivar incluso en la muerte.
En contraste, T. b. rhodesiense, presente en África oriental y meridional, es responsable de las infecciones agudas con un rápido inicio sumado a una progresión más veloz de la enfermedad.
Todos los tripanosomiasis infecciosas humanas son transmitidos por la mosca tse-tsé (Glossina sp) en una región claramente definida de África, llamada tse-tsé belt (2).
La enfermedad se limita a la zona del África subsahariana y se solapa con la distribución de la mosca tse-tsé, que actúa como vector.
La epidemiología de THA es heterogénea, caracterizándose por dos etapas definidas. La primera etapa implica la presencia de tripanosomas en el sistema hemo-linfático. En este momento, los síntomas son bastante inespecíficos, incluyendo malestar general, fiebre, y dolor de cabeza. La segunda etapa comienza una vez que los parásitos llegan al cerebro y provocan alteración de las funciones neurológicas, incluyendo cambios psicológicos y trastornos del sueĖo, lo que da lugar al término comúnmente empleado para definir la THA, "enfermedad del sueĖo"(3).
La realidad es que THA es todavía una enfermedad olvidada que requiere mayor investigación en la prevención, diagnóstico y estrategias de tratamiento. En las últimas décadas, la THA ha vuelto a ser una enfermedad reemergente, volviendo a la crítica situación vivida a principios del siglo XX. En el aĖo 2000, se calcula que 300.000 personas estaban infectadas. Sin embargo, ya que sólo el 10-15% de los 60 millones de personas que viven en zonas de riesgo están bajo control, se puede considerar que el número de personas infectadas está probablemente muy subestimada (1).
El tratamiento de esta enfermedad es complejo. Si analizamos el arsenal terapéutico frente a la THA, podemos observar como los fármacos empleados requieren generalmente suministración por vía parenteral, lo que dificulta la adhesión y accesibilidad del paciente al tratamiento.
Además de lo citado anteriormente, el tratamiento actual presenta problemas de eficacia y seguridad (3).
Por todo lo expuesto, se genera una necesidad real de encontrar un mejor y eficaz tratamiento para contener los brotes de infección a través de métodos de búsqueda económicamente sostenibles.
Uno de los métodos que reúnen este requisito de sostenibilidad y eficacia son las técnicas “in silico” en el campo de la investigación científica. La introducción de ordenadores cada vez más eficientes ha permitido lograr grandes avances en el campo bioinformático, hasta el punto de que hoy en día estas técnicas representan una herramienta básica en el campo de la investigación (4).
Los llamados estudios de relación cuantitativa estructura-actividad o quantitative structure–activity relationship (QSAR) pueden reducir los costosos fallos en la selección de compuestos candidatos a fármacos en los ensayos clínicos, mediante el filtrado de bibliotecas combinatorias.
El cribado virtual o virtual screening permite identificar a los compuestos que presentan toxicidad y/o malas características farmacocinéticas (5), al principio de la fase de desarrollo del fármaco.
Por último, aplicando esta metodología se logra cribar los muchos compuestos que suelen mostrar actividad inespecífica en varios ensayos y que rara vez resultan ser verdaderamente eficaces o activos (6).
En la búsqueda de relaciones cuantitativas estructura-actividad, la topología molecular (TM), se ha convertido en la actualidad en una herramienta muy útil para caracterizar estructuralmente a una molécula y por tanto predecir de forma rápida y precisa diversas propiedades fisicoquímicas y biológicas de la misma (7-14).
Con ayuda de TM las moléculas son caracterizadas estructuralmente a través de los índices topológicos (IT) (15). Estos IT son capaces de caracterizar las propiedades fisicoquímicas y biológicas de una determinada molécula (tamaĖo molecular, presencia de ciclos, ramificaciones…etc.). El cálculo de IT no es complejo, y tiene muchas ventajas. Una de las principales ventajas, reside en el hecho de que los descriptores topológicos no están atados a su disposición dentro de la molécula. Es decir, son valores invariables de la molécula y no depende de la conformación de la misma en un determinado momento.
Por medio de diversos enfoques estadísticos llegamos a la obtención de un modelo topológico, es decir una ecuación matemática basada en estos IT para una determinada propiedad físico-química o biológica. Después de la obtención del modelo topológico para la predicción de una determinada actividad farmacológica, un cribado virtual, se lleva a cabo con el fin de rastrear e identificar nuevos compuestos con la actividad farmacológica deseada (15, 16).
Los índices topológicos se han utilizado para la predicción de diversas propiedades fisicoquímicas y biológicas en grupos de compuestos con una amplia diversidad estructural (15,17).
El método topológico ha sido aplicado por la “Unidad de DiseĖo de Fármacos y Conectividad Molecular” de la Universitat de Valencia, para la búsqueda de nuevos compuestos anti-protozoarios (18), anti-maláricos (19, 20), los anti-microbianos (21, 22), anti-fúngicos (23), anti-neoplásicos (24), agentes broncodilatadores (25, 26), citostáticos (27), anti-inflamatorios (7,16,28), anti-virales VIH-1 (29), compuestos activos frente a Colitis ulcerosa (30) y compuestos antihistamínicos (31).
Varias patentes se han derivado del empleo de TM para la búsqueda de nuevos fármacos en el tratamiento de la malaria (20), cáncer de pulmón (32, 33), y la enfermedad de Alzheimer por nuestro grupo de investigación (34).
En cuanto al estado actual del desarrollo clínico de nuevos compuestos frente a THA, cabe destacar la aparición en escena del compuesto fexinidazol. Este compuesto es un 5-nitroimidazol activo frente al T. b. gambiense y al T. b. rhodesiense, presentando un perfil de seguridad favorable además de ser suministrado por vía oral (35).
Los nitro-imidazoles son derivados conocidos por su actividad antibacteriana y anti-protozoaria. En el trabajo de Bourdin B. et al. se describe la actividad de 49 compuestos derivados del 1-aril-4-nitro-1H-imidazol frente a tripanosomiasis africana aguda y crónica en modelos experimentales en ratones (36).
El objetivo del presente trabajo, es la elaboración de un modelo topológico-matemático encaminado a buscar nuevos compuestos derivados del 1-aril-4-nitro-1H-imidazol con potencial actividad anti-tripanosómica.
2. MATERIAL Y MÉTODOS
Compuestos analizados
Para este estudio se seleccionaron un grupo de 49 compuestos derivados del 1-aril-4-nitro-1H-imidazol recogidos en el trabajo publicado por Bourdin et al., los cuales mostraron cierta actividad anti-tripanosómica (36).
La Tabla 1 recoge la estructura química y la actividad antiparasitaria frente a Trypanosoma en términos de concentración inhibitoria 50, CI50 (ĶM) para cada compuesto (la numeración seguida es la misma que aparece en el trabajo original) Bourdin et al. (36).
Descriptores topológicos empleados
Los índices topológicos empleados en este trabajo fueron los siguientes: índices de conectividad tipo Randic (37) y Kier-Hall (38) hasta el orden cuarto (mχt, mχtv ) e índices topológicos de carga, ICT (39) hasta el orden quinto, (Jm, Gm, Jmv , Gmv ). En total, cada compuesto fue caracterizado con una serie de 62 índices. Todos los descriptores han sido obtenidos gracias al programa DESMOL11 (disponible vía e-mail).
Tabla1.- Estructura química, actividad experimental (CI50 (μM) ), valor de los descriptores presentes en FD así como la clasificación obtenida para cada compuesto analizado en base a la función discriminante seleccionada.
Compuesto |
Sustituyente |
CI50expa |
4χpc |
G2v |
J5 |
Clasexp |
Prob(act)b |
FDb |
Clascalcb |
1-aril-4-nitro-2H-imidazoles monosustituidos en el anillo benzénico |
|||||||||
1 |
H |
4.49 |
1.0739 |
3.6631 |
0.0396 |
A |
0.994 |
5.10 |
A |
2 |
2-F |
2.85 |
1.3700 |
4.1082 |
0.0553 |
A |
0.476 |
-0.09 |
I |
3 |
3-F |
0.72 |
1.2460 |
3.7370 |
0.0407 |
A |
0.998 |
6.01 |
A |
4 |
4-F |
2.53 |
1.2663 |
3.9592 |
0.0447 |
A |
0.981 |
3.92 |
A |
5 |
2-Cl |
n.a. |
1.3700 |
4.3677 |
0.0553 |
I |
0.319 |
-0.76 |
I |
6 |
3-Cl |
0.72 |
1.2460 |
3.9965 |
0.0407 |
A |
0.995 |
5.35 |
A |
7 |
4-Cl |
0.72 |
1.2663 |
4.2187 |
0.0447 |
A |
0.963 |
3.26 |
A |
8 |
2-Br |
n.a. |
1.3700 |
4.4310 |
0.0553 |
I |
0.285 |
-0.92 |
I |
9 |
3-Br |
0.9 |
1.2460 |
4.0598 |
0.0407 |
A |
0.994 |
5.19 |
A |
10 |
4-Br |
0.97 |
1.2663 |
4.2820 |
0.0447 |
A |
0.957 |
3.10 |
A |
11 |
3-NO2 |
0.77 |
1.6959 |
4.1582 |
0.0512 |
A |
0.990 |
4.56 |
A |
12 |
3-CF3 |
1.45 |
2.2723 |
5.2190 |
0.0621 |
A |
0.924 |
2.51 |
A |
13 |
4-CF3 |
0.7 |
2.2861 |
5.4413 |
0.0621 |
A |
0.888 |
2.07 |
A |
14 |
2-CF3O |
20.76 |
1.8537 |
4.1339 |
0.0789 |
I |
0.003 |
-5.84 |
I |
15 |
3-CF3O |
1.76 |
1.7827 |
3.7626 |
0.0614 |
A |
0.882 |
2.01 |
A |
16 |
4-CF3O |
0.16 |
1.7992 |
3.9848 |
0.0614 |
A |
0.832 |
1.60 |
A |
17 |
3-Me |
1.82 |
1.2460 |
4.9965 |
0.0407 |
A |
0.942 |
2.80 |
A |
18 |
4-Me |
11.96 |
1.2663 |
5.2187 |
0.0447 |
I |
0.670 |
0.71 |
A |
19 |
3-MeO |
1.92 |
1.3113 |
4.0190 |
0.0463 |
A |
0.970 |
3.48 |
A |
20 |
4-MeO |
3.19 |
1.3278 |
4.2412 |
0.0500 |
A |
0.812 |
1.47 |
A |
21 |
4-CO2Bu |
28.03 |
1.5207 |
7.8516 |
0.0583 |
I |
0.000 |
-9.54 |
I |
1-aril-4-nitro-2H-imidazoles disustituidos en el anillo benzénico |
|||||||||
22 |
2,3-diF |
2.18 |
1.7549 |
4.4043 |
0.0554 |
A |
0.937 |
2.71 |
A |
23 |
2,4-diF |
n.a. |
1.5384 |
4.1821 |
0.0591 |
I |
0.419 |
-0.33 |
I |
24 |
2,5-diF |
1.64 |
1.5431 |
4.1821 |
0.0554 |
A |
0.788 |
1.31 |
A |
25 |
2,6-diF |
n.a. |
1.6255 |
4.3311 |
0.0690 |
I |
0.015 |
-4.19 |
I |
26 |
3,4-diF |
0.76 |
1.6773 |
4.2552 |
0.0454 |
A |
0.999 |
6.66 |
A |
27 |
3,5-diF |
0.49 |
1.3923 |
3.5886 |
0.0417 |
A |
0.999 |
7.32 |
A |
28 |
2,3-diCl |
1.9 |
1.7549 |
4.9233 |
0.0554 |
A |
0.799 |
1.38 |
A |
29 |
2,4-diCl |
n.a. |
1.5384 |
4.7011 |
0.0591 |
I |
0.161 |
-1.65 |
I |
30 |
2,5-diCl |
n.a. |
1.5431 |
4.7011 |
0.0554 |
I |
0.497 |
-0.01 |
I |
31 |
3,4-diCl |
0.1 |
1.6773 |
4.7742 |
0.0454 |
A |
0.995 |
5.33 |
A |
32 |
3,5-diCl |
0.7 |
1.3923 |
4.1076 |
0.0417 |
A |
0.998 |
5.99 |
A |
33 |
3-Cl-4-F |
0.7 |
1.6773 |
4.5147 |
0.0454 |
A |
0.998 |
6.00 |
A |
34 |
2-Br-4-Me |
n.a. |
1.5384 |
5.7644 |
0.0591 |
I |
0.013 |
-4.36 |
I |
35 |
3-Me-4-Br |
0.99 |
1.6773 |
5.8375 |
0.0454 |
A |
0.932 |
2.62 |
A |
36 |
3-Br-4-Me |
1.99 |
1.6773 |
5.8375 |
0.0454 |
A |
0.932 |
2.62 |
A |
37 |
2,4-diMe |
n.a. |
1.5384 |
6.7011 |
0.0591 |
I |
0.001 |
-6.75 |
I |
38 |
3,4-diMe |
2.12 |
1.6773 |
6.7742 |
0.0454 |
A |
0.557 |
0.23 |
A |
39 |
2,4-diMeO |
n.a. |
1.6114 |
4.7461 |
0.0733 |
I |
0.001 |
-7.24 |
I |
Compuesto |
Sustituyente |
CI50expa |
4χpc |
G2v |
J5 |
Clasexp |
Prob(act)b |
FDb |
Clascalcb |
40 |
3,4-diMeO |
n.a. |
1.6427 |
4.8193 |
0.0548 |
I |
0.704 |
0.87 |
A |
41 |
3,4-O2-CH2 |
0.94 |
1.5829 |
4.7359 |
0.0376 |
A |
1.000 |
7.93 |
A |
42 |
3-CO2Bu-4-Cl |
141.32 |
1.7803 |
8.4071 |
0.0599 |
I |
0.000 |
-9.21 |
I |
43 |
3-CO2H-4-Cl |
95.54 |
1.7689 |
7.5295 |
0.0515 |
I |
0.030 |
-3.47 |
I |
1-aril-2-metil-4-nitro-imidazoles mono o disustituidos en el anillo benzénico |
|||||||||
44 |
4-CF3O |
11.89 |
2.1018 |
6.3913 |
0.0643 |
I |
0.046 |
-3.02 |
I |
45 |
4-CF3 |
11.28 |
2.5887 |
7.8478 |
0.0652 |
I |
0.068 |
-2.61 |
I |
46 |
2-Br-4-Me |
124.6 |
1.8430 |
8.1709 |
0.0627 |
I |
0.000 |
-9.27 |
I |
47 |
3-Cl-4-F |
27.03 |
1.9799 |
6.9212 |
0.0534 |
I |
0.311 |
-0.79 |
I |
48 |
4-Br-3-Me |
20.16 |
1.9799 |
8.2440 |
0.0534 |
I |
0.015 |
-4.17 |
I |
49 |
3-Br-4-Me |
23.54 |
1.9799 |
8.2440 |
0.0534 |
I |
0.015 |
-4.17 |
I |
Fármacos de referencia |
|||||||||
Fexinidazol |
2.57 |
||||||||
Megazol |
0.1 |
||||||||
Melarsoprol |
0.009 |
||||||||
Eflornitine |
3.8 |
||||||||
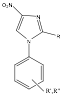
a Datos de actividad anti-tripanosómica CI50 experimental obtenidos del trabajo publicado por Bourdin et al. (36). b Obtenido a partir de la ecuación 1. c n.a.: no presenta actividad anti-tripanosómica. Estructura base:
Algoritmo QSAR: Análisis lineal discriminante
La búsqueda de un modelo topológico-matemático capaz de identificar compuestos con actividad anti-tripanosómica, se ha realizado aplicando la técnica estadística del análisis lineal discriminante ALD, por medio de la cual, es posible obtener funciones lineales capaces de clasificar los compuestos estudiados en categorías o grupos y discriminar entre compuestos activos e inactivos (15).
En nuestro caso, el ALD se ha aplicado a dos series de compuestos:
La primera serie formada por compuestos con probada actividad inhibitoria frente a Trypanosoma (en nuestro caso, compuestos con CI50<5μM) y la segunda, por compuestos considerados inactivos (CI50>5μM).
La capacidad discriminante fue expresada por medio del porcentaje de clasificación correcta para cada grupo. El ALD se realizó con la ayuda del paquete estadístico StatSoft STATISTICA, versión 8.0. (40) La selección de los índices en la función discriminante se basa en el parámetro F-Snedecor, y el criterio de clasificación empleado es la menor distancia de Mahalanobis. La calidad de la función de discriminación se evalúa con el parámetro lambda de Wilks empleando el test de igualdad de las medias de grupo para las variables de la función de discriminación (41-42).
La razón principal por la cual hemos escogido la técnica ALD, se debe a que de 10 de los 49 compuestos estudiados (20 % de los compuestos estudiados) no disponíamos del valor de CI50 y mediante el ALD se pueden tratar valores discretos de la propiedad a diferencia de lo que ocurre con otras técnicas como puede ser la regresión multilineal (43).
Diagramas de distribución de la actividad farmacológica, DDAF
Una vez obtenida la función discriminante, es interesante realizar el correspondiente diagrama de distribución de la actividad farmacológica (DDAF), para cada una de ellas.
Estos gráficos son útiles para determinar el intervalo de la función discriminante en el que la expectativa, E, o probabilidad de encontrar compuestos activos, es máxima. Los DDAF son histogramas en los que se representa en ordenadas la E y en el eje de abscisas, el valor de la función discriminante, FD. Para un intervalo arbitrario de FD, se puede definir la expectativa de actividad, Ea, como: Ea = a/(i+1), en donde a representa el número de compuestos activos en el intervalo dividido por el número total de compuestos activos, e i representa el número de compuestos inactivos en el intervalo dividido por el número total de compuestos inactivos.
La probabilidad de inactividad viene definida de una manera simétrica como Ei=i/(a+1). Esta representación nos da una buena visualización de las regiones de mínimo solapamiento, y permite la selección de intervalos de FD en los que la probabilidad de encontrar compuestos activos es máxima (44).
Análisis de validación
La calidad predictiva y robustez del modelo de predicción seleccionado debe evaluarse por medio de un test de validación interna. En este caso se han adoptado dos estrategias:
• La primera validación ha consistido en aplicar el método Jack-knife (45), el cual puede definirse como un procedimiento estadístico de re-muestreo para estimar el error estándar de una magnitud. Este método consiste en extraer un compuesto de la serie y volver a calcular el modelo utilizando como conjunto de entrenamiento N-1 compuestos de forma que la propiedad es entonces predicha para el elemento eliminado. Este proceso se repite para todos los compuestos de la serie, obteniendo una predicción para cada uno.
• La segunda validación o validación cruzada, consiste en dividir la data en varios subgrupos (5 en nuestro caso con 11 compuestos cada uno). Cuatro de los subgrupos formarán el grupo de entrenamiento y el quinto el grupo test. El proceso se repite cinco veces cambiando los subgrupos de entrenamiento y test. La división de los subgrupos fue llevada a cabo aleatoriamente.
El objetivo de este test de validación cruzada es determinar la robustez del modelo, garantizando que el porcentaje de acierto del modelo no depende exclusivamente de unos pocos compuestos empleados para su elaboración, sino que los compuestos que conforman la data tienen similar peso a la hora de elaborar el modelo.
3. RESULTADOS y discusión
El análisis de discriminación lineal se realizó con los 49 compuestos que formaban la data y los 62 descriptores topológicos que describen los aspectos topológicos y estructurales de cada molécula.
Tabla 2.- Matriz de clasificación obtenida con la función discriminante seleccionada y el test de validación cruzada.
Grupo de entrenamiento |
Grupo test |
|||||
FD |
l |
% Activos |
% Inactivos |
% Activos |
% Inactivos |
|
(lambda de Wilks) |
||||||
Modelo seleccionado |
0.349 |
100 |
86 |
- |
- |
|
CV1 |
0.292 |
100 |
88 |
100 |
60 |
|
CV2 |
0.309 |
100 |
81 |
83 |
80 |
|
CV3 |
0.376 |
95 |
88 |
100 |
80 |
|
CV4 |
0.368 |
100 |
81 |
100 |
80 |
|
CV5 |
0.330 |
100 |
88 |
100 |
60 |
|
Media CV |
0.335 |
99 |
85 |
97 |
72 |
|
La separación del grupo activo y del inactivo, se hizo en base al valor de CI50=5mM. La función discriminante seleccionada está formada por los descriptores 4cpc, G2v y J5 (evaluando el primero el grado de ramificación de cada compuesto y los dos restantes la distribución de carga intramolecular).
FD = 21,54 + 9,26 4χpc - 2,55 G2V – 430,62 J5 Ecuación 1
N=49; F=28,007; l(Wilks’ lambda)=0,349
En base a esta función, un compuesto será clasificado como activo “A” si presenta un valor de FD>0, en caso contrario, será catalogado como inactivo “I” (ver Tabla 1).
La matriz de clasificación obtenida para el modelo matemático-topológico seleccionado (ver Tabla 2), es muy significativa: el 100% de los compuestos activos (28 de 28 compuestos) son clasificados correctamente, así como el 85,7% del grupo inactivo (18 de 21 compuestos) lo que representa un porcentaje de acierto medio del 93%.
El modelo fue sometido a dos análisis de validación interna: método de Jack-knife o leave-one-out, así como una cross-validation o validación cruzada. El primero tal y cómo explicamos en párrafos anteriores, consistió en una validación interna con pérdida de un compuesto y posterior predicción del mismo por el modelo.
La validación del modelo siguiendo el método de Jack-knifed, para el grupo de entrenamiento, muestra una matriz de clasificación en la que 26 de 28 compuestos activos son clasificados correctamente (93%) y 18 de 21 compuestos inactivos fueron correctamente dispuestos por el modelo (86%). Obteniendo por tanto, un porcentaje de correcta clasificación global del 90 %. Por lo que la FD (Ec.1) para la búsqueda de compuestos con actividad anti-tripanosómica supera claramente este primer test de validación interna, demostrando ser un modelo robusto.
Asimismo, se realizó una segunda prueba de validación interna al modelo (Ec.1).
Para llevar a cabo este test de validación cruzada, se dividió el grupo estudiado en cinco subgrupos (CV1-CV5): serie CV1 (1, 5, 7, 13, 20, 21, 28, 30, 36, 40, 45), serie CV2 (2, 8, 9, 15, 22, 23, 31, 34, 38, 42, 46), serie CV3 (3, 10, 14, 16, 24, 25, 32, 37, 41, 43, 47), serie CV4 (4, 11, 17, 18, 26, 29, 33, 39, 44, 48), serie CV5 (6, 12, 19, 21, 27, 30, 35, 40, 45, 49). Estas series corresponden en el estudio de validación cruzada al grupo de test (es decir, no participan en la elaboración del modelo), mientras que los compuestos restantes se asignan al grupo de entrenamiento.
En la Tabla 2, se muestran los valores de λ (lambda de Wilks) y la matriz de clasificación para los compuestos pertenecientes tanto al grupo de entrenamiento como al grupo test.
La variabilidad de λ es pequeĖa para cada serie y el valor promedio de λ tras cinco ensayos de validación cruzada, es muy similar al obtenido con el modelo seleccionado (0,349). Por lo que podemos afirmar que el modelo seleccionado para la búsqueda de compuestos con actividad anti-tripanosómica es robusto.
La Figura 1, muestra el diagrama de distribución de la actividad anti-trypanosómica obtenido a partir del análisis lineal discriminante realizado.
Las barras negras y blancas corresponden con el grupo activo e inactivo, respectivamente. Como puede verse, los compuestos activos se reagrupan preferentemente dentro del intervalo de la FD comprendido entre -1,5 y 8. Por tanto, un compuesto activo mostrará un valor de FD preferiblemente dentro de ese intervalo. También puede observarse en el diagrama que prácticamente no se aprecia solapamiento entre compuestos activos e inactivos con valores de FD>0.

Figura 1.- Diagrama de distribución de la actividad anti-trypanosómica obtenido tras el análisis lineal discriminante realizado.
Por último, se realizó un cribado molecular virtual encaminado a buscar nuevas estructuras químicas derivadas del 4-nitro-imidazol que pudiesen ser potencialmente activas frente a T. b. rhodesiense. Se dibujaron 21 moléculas y se les aplicó el modelo discriminante seleccionado.
La Tabla 3 muestra los resultados obtenidos. Como puede observarse, 10 de las 21 moléculas analizadas se pueden considerar potencialmente activas.
El siguiente paso a realizar sería sintetizarlas u obtenerlas vía comercial y proceder a ensayarlas en el laboratorio para comprobar la actividad predicha por el modelo discriminante aplicado.
La principal ventaja del método topológico seguido sería el ahorro en tiempo y dinero invertido en la búsqueda de nuevos compuestos activos frente a T. b. rhodesiense.
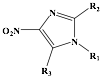
Tabla 3.- Resultados obtenidos del cribado molecular realizado con nuevos derivados del 4-nitro-imidazol utilizando el análisis lineal discriminante.
N0. |
R1 |
R2 |
R3 |
4χpc |
G2v |
J5 |
Prob.(A) |
FD |
Clascalc |
1 |
CH3 |
H |
Ph |
1.345 |
6.182 |
0.041 |
0.614 |
0.47 |
A |
2 |
|
H |
H |
1.577 |
6.720 |
0.042 |
0.755 |
1.13 |
A |
3 |
|
H |
H |
1.677 |
4.515 |
0.045 |
0.998 |
5.99 |
A |
4 |
|
H |
H |
1.342 |
4.552 |
0.041 |
0.990 |
4.62 |
A |
5 |
|
H |
H |
1.342 |
4.552 |
0.039 |
0.995 |
5.39 |
A |
6 |
H |
H |
Ph |
1.369 |
6.182 |
0.044 |
0.359 |
-0.57 |
I |
7 |
H |
|
H |
1.879 |
7.747 |
0.031 |
0.997 |
5.70 |
A |
8 |
|
H |
H |
1.374 |
4.390 |
0.064 |
0.012 |
-4.37 |
I |
9 |
|
H |
H |
1.543 |
4.182 |
0.055 |
0.788 |
1.31 |
A |
10 |
|
H |
H |
1.625 |
4.850 |
0.069 |
0.004 |
-5.51 |
I |
11 |
|
H |
H |
1.543 |
4.701 |
0.055 |
0.497 |
-0.01 |
I |
12 |
|
H |
H |
1.755 |
4.664 |
0.055 |
0.885 |
2.04 |
A |
13 |
|
H |
H |
1.538 |
5.764 |
0.059 |
0.013 |
-4.36 |
I |
14 |
|
CH3 |
H |
2.085 |
6.169 |
0.067 |
0.020 |
-3.87 |
I |
N0. |
R1 |
R2 |
R3 |
4χpc |
G2v |
J5 |
Prob.(A) |
FD |
Clascalc |
15 |
|
CH3 |
H |
2.575 |
7.626 |
0.065 |
0.102 |
-2.17 |
I |
16 |
|
H |
H |
1.707 |
4.456 |
0.071 |
0.010 |
-4.61 |
I |
17 |
|
H |
H |
1.711 |
4.380 |
0.055 |
0.933 |
2.64 |
A |
18 |
|
H |
H |
2.228 |
5.590 |
0.077 |
0.004 |
-5.45 |
I |
19 |
|
H |
H |
1.370 |
5.368 |
0.055 |
0.035 |
-3.31 |
I |
20 |
|
CH3 |
H |
2.013 |
6.787 |
0.058 |
0.090 |
-2.31 |
I |
21 |
|
H |
H |
1.074 |
4.181 |
0.040 |
0.978 |
3.78 |
A |
Estructura base:
4. conclusioneS
Los resultados obtenidos en este trabajo, ponen de manifiesto la eficacia de la topología molecular en la predicción de la actividad anti-tripanosómica.
A través de este método topológico se pueden buscar compuestos con actividad frente a T. b. rhodesiense mediante la aplicación de una función discriminante (Ecuación 1), capaz de diferenciar compuestos activos de inactivos. La función discriminante obtenida se ha aplicado a otros compuestos estructuralmente similares, a modo de cribado virtual para la búsqueda de nuevos compuestos potencialmente activos frente a T. b. rhodesiense.
De forma que podemos decir que la topología molecular es eficaz en el descubrimiento de agentes terapéuticos para esta enfermedad que afecta a gran parte de África.
Aplicando el método topológico, podemos descubrir nuevas moléculas con teórica actividad anti-tripanosómica, con un coste mínimo y optimizar las probabilidades de éxito con nuevos tratamientos frente a T. b. rhodesiense.
5. agradecimientos
Este trabajo ha sido realizado gracias al apoyo del Ministerio de Economía y Competitividad, EspaĖa (proyecto SAF2009-13059-C03-02). El presente estudio se deriva de la colaboración de la “Unidad de diseĖo de fármacos y conectividad molecular” perteneciente al departamento de Química-física de Facultad de Farmacia (Universitat de Valencia) con el Master Internacional en Enfermedades Parasitarias Tropicales (curso 2011/2012).
Una de los autores, M. Galvez Llompart es beneficiaria del programa “Atracció de talent” de formación de personal investigador de carácter predoctoral concedido por la Universitat de Valencia.
6. REFERENCIAS
1. Geigera A., Simob G., Grébauta P., Peltierc J.B., Cunya G., Holzmullera P. (2011). Transcriptomics and proteomics in human African trypanosomiasis: Current status and perspectives J. Proteomics 74(9), 1625-1643.
2. La Greca F., & Magez S. (2011). Vaccination against trypanosomiasis. Can it be done or is the trypanosome truly the ultimate immune destroyer and escape artist? Hum Vaccin 7(11), 1225-1233.
3. Brun R., Don R., Jacobs R.T., Wang M.Z., Barrett M.P. (2011). Development of novel drugs for human African trypanosomiasis. Future Microbiol 6(6), 677-691.
4. Hansch C., & Fujita T. (1964) p-tau-pi analysis. A method for the correlation of biological activity and chemical structure. J Am Chem Soc 86(8), 1616-1626.
5. van de Waterbeemd H., & Gifford E. (2003) ADMET in silico modelling: towards prediction paradise? Nat Rev Drug Discov 2(3), 192-204.
6. Roche O., Schneider P., Zuegge J., et al. (2002) Development of a virtual screening method for identification of “frequent hitters” in compound libraries. J Med Chem 45(1), 137-142.
7. Galvez-Llompart M., Giner M., Recio C., et al.(2010) Application of molecular topology to the search of novel NSAIDs: experimental validation of activity. Lett Drug Des Discov 7(6),438-445.
8. de Julian-Ortiz J.V., Galvez J., Munoz-Collado C., et al.(1999) Virtual combinatorial syntheses and computational screening of new potential anti-herpes compounds. J Med Chem 42(17),3308-3314.
9. Bruno-Blanch L., Galvez J., Garcia-Domenech R.(2003) Topological virtual screening: A way to find new anticonvulsant drugs from chemical diversity. Bioorg Med Chem Lett 13(16), 2749-2754.
10. Rios-Santamarina I., Garcia-Domenech R., Galvez J. (1998) New bronchodilators selected by molecular topology. Bioorg Med Chem Lett 8(5),477-482.
11. Galvez J., Garcia-Domenech R., de Julian-Ortiz J., et al. (1995) Topological approach to drug design. J Chem Inf Comput Sci 35(2), 272-284.
12. Duart M., Garcia-Domenech R., Anton-Fos G., et al. (2001) Optimization of a mathematical topological pattern for the prediction of antihistaminic activity. J Comput Aided Mol Des 15(6), 561-572.
13. Galvez J., Garcia-Domenech R., Gomez-Lechon M., et al. (2000) Use of molecular topology in the selection of new cytostatic drugs. J Mol Struct Theochem 504(1-3), 241-248.
14. de Gregorio Alapont C., Garcia-Domenech R., Galvez J., et al.(2000) Molecular topology: A useful tool for the search of new antibacterials. Bioorg Med Chem Lett 10(17), 2033-2036.
15. Gálvez J., Gálvez-Llompart M., García-Domenech R.(2012) Molecular topology as a novel approach for drug discovery. Expert Opin Drug Discov 7(2), 133-153.
16. Galvez-Llompart M., Zanni R., Garcia-Domenech R. (2011) Modeling Natural Anti-Inflammatory Compounds by Molecular Topology. Int J Mol Sci 12(12), 9481–9503.
17. García-Doménech R., Gálvez J., Julián-Ortiz J.V., Pogliani L. (2008) Some new trends in chemical graph theory. Chem Rev 108(3), 1127-1169.
18. Garcia-Domenech R., Domingo-Puig C., Esteve-Martinez M.A., Schmitt J., Vera-Martinez J., Chindemi A., Galvez J. (2008) Aplicacion de la Topologia Molecular en la busqueda de nuevos agentes activos frente a Leishmania. An. R. Acad. Nac. Farm. 74, 345-367.
19. Mahmoudi N., de Julian-Ortiz J.V., Ciceron L., et al.(2006) Identification of new antimalarial drugs by linear discriminant analysis and topological virtual screening. J Antimicrob Chemother 57(3), 489-97.
20. Mahmoudi N., Garcia-Domenech R., Galvez J., et al.(2008) New active drugs against liver stages of plasmodium predicted by Molecular Topology. Antimicrob Agents Chemother 52(4), 1215-1220.
21. de Gregorio Alapont C., Garcia-Domenech R., Galvez J., et al. (2000) Molecular Topology: A useful tool for the search of new antibacterials. Bioorg Med Chem Lett 10(17), 2033-2036.
22. Mishra R.K., Garcia-Domenech R., Galvez J. (2001) Getting discriminant functions of antibacterial activity from physicochemical and topological parameters. J Chem Inf Comput Sci 41(2), 387-393.
23. Pastor L., Garcia-Domenech R., Galvez J., et al. (1998) New antifungals selected by Molecular Topology. Bioorg Med Chem Lett 8(18), 2577-2582.
24. Galvez J., García-Domenech R., de Julian-Ortiz J.V. (1998) Design of new antineoplastic lead drugs by molecular topology. Expert Opin Drug Discov 2(2), 265-268.
25. Rios-Santamarina I., Garcia-Domenech R., Galvez J.(1998) New bronchodilators selected by Molecular Topology. Bioorg Med Chem Lett 8(5),477-482.
26. Rios-Santamarina I., Garcia-Domenech R., Galvez J., et al.(2004) Getting new bronchodilator compounds from Molecular Topology. Eur J Pharm Sci 4, 271−277.
27. Galvez J., Garcia-Domenech R., Gomez-Lechon M., et al. (2000) Use of Molecular Topology in the selection of new cytostatic drugs. J Mol Struct Theochem 504(1-3), 241-248.
28. Pla-Franco J., Gálvez-Llompart M., Gálvez J., García-Domenech R. (2011) Application of Molecular Topology for the Prediction of Reaction Yields and Anti-Inflammatory Activity of Heterocyclic Amidine Derivatives. Int J Mol Sci 12, 1281-1292.
29. Garcia-Domenech R., Montealegre M.C., Nagham E.G., Sandoval N., Santana M., Galvez J. (2010) Aplicacion de la topologia molecular para la prediccion de la actividad anti-VIH-1 de un grupo de compuestos analogos del acyclovir y ganciclovir. An. R. Acad. Nac. Farm 76(1), 45-57.
30. Gálvez-Llompart M., Recio M.C., García-Domenech R. (2011) Topological virtual screening: a way to find new compounds active in ulcerative colitis by inhibiting NF-κB. Mol Divers 15(4), 917-26.
31. Casabán-Ros E., Antón-Fos G.M., Gálvez J., et al. (1999) Search for New Antihistaminic Compounds by Molecular Connectivity. Quant Struct-Act Relat 18, 35-42.
32. Jasinski P., Welsh B., Galvez J., et al. (2008) A novel quinoline, MT477: Suppresses cell signaling through ras molecular pathway, inhibits PKC activity, and demonstrates in vivo anti-tumor activity against human carcinoma cell lines. Invest New Drugs 26(3),223-232.
33. Jasinski P., Zwolak P., Isaksson V.R., et al. (2011) MT103 inhibits tumor growth with minimal toxicity in murine model of lung carcinoma via induction of apoptosis. Invest New Drugs 29(5),1-7.
34. Galvez J., Llompart J., Land D., et al. (2010) Mount Sinai School of Medicine of New York University, USA, Medisyn Technologies I, assignees. Compositions for treatment of Alzheimer’s disease using Abeta-reducing and/or Abeta-anti-aggregation compounds. WO/2010/114636; PCT/US2010/001041.
35. Lutje V., Seixas J., Kennedy A.(2010) Chemoterapy for second-stage Human African trypanosomiasis. Cochrane Database of Systematic Reviews. Issue 8. Art. No.: CD006201. DOI:10.1002/14651858.CD006201.
36. Bourdin B., Jedrysiak R., Tweats D., Brun R., Kaiser M., Suwinski J., Torreele E. (2011)1-Aril-4-nitro-1H-imidazoles, a new promising series for the treatment of human African trypanosomiasis. Eur J Med Chem 46, 1524-1535.
37. Randic, M. (1975) On characterization of molecular branching. J Am Chem Soc 97, 6609-6615.
38. Kier L.B., Hall L.H. (1986) Molecular Connectivity in Structure-Activity Analysis. Research Studies Press, Letchworth, UK.
39. Gálvez J., García-Doménech R., Salabert M.T., Soler R. (1994) Charge Indices. New topological descriptors. J Chem Inf Comput Sci 34, 520-525.
40. Statistica (data analysis software system), version 9.0(2009) Statsoft: Tulsa, OK.
41. Wold S., Eriksson L., Clementi S. (2008) Statistical validation of QSAR results, in Chemometric Methods in Molecular Design (ed H. van de Waterbeemd), Wiley-VCH Verlag GmbH, Weinheim, Germany. doi: 10.1002/9783527615452.ch5.
42. Dixon W.J. (1990) BMDP statistical software manual: to accompany the 1990 software release. Univ of California Pr; San Francisco.
43. Roy P.P., Leonard J.T., Roy K. (2008) Exploring the impact of size of training sets for the development of predictive QSAR models. Chem Intell Lab Syst 90(1), 31-42.
44. Galvez J., Garcia-Domenech R., de Gregorio Alapont C., et al. (1996) Pharmacological distribution diagrams: a tool for de novo drug design. J Mol Graph 14(5), 272-276.
45. Lachenbruch P.A., Mickey M.R. (1968) Estimation of error rates in discriminant analysis. Technometrics 10(1), 1-11.